Análisis transcriptómico en frutales del género Prunus: Aplicaciones a los programas de mejora
En los últimos años se han desarrollado una serie de herramientas de análisis secuenciación de alto rendimiento o masiva de RNA (transcriptoma) lo suficientemente económicas y precisas para ser aplicadas en los programas de mejora genética de frutales del género Prunus. Estas técnicas permiten identificar de forma precisa los genes responsables de características agronómicas deseables como la resistencia a virus, floración, o calidad del fruto. Además la existencia de genomas completos de especies frutales está sirviendo de referencia para el análisis de los datos obtenidos mediante secuenciación masiva de transcriptomas y la posterior identificación de estos genes responsables de los caracteres de interés. Por otro lado, la secuenciación masiva de transcriptomas (RNA-Seq) está ayudando a tener un mejor conocimiento de los procesos biológicos y moleculares asociados a la expresión de estos caracteres de interés agronómico. Una vez identificados estos genes, podemos utilizar esta información para seleccionar las plántulas portadoras de esas características mediante el uso de la técnica RT-PCR cuantitativa (qPCR) con una aplicación potencial en la selección asistida por marcadores.

Importancia económica de los frutales de hueso y programas de mejora genética públicos en España
El género Prunus (dentro de la familia Rosaceae y el orden Rosales) incluye más de 230 especies de las cuales se cultivan principalmente nueve de ellas dentro de los tres subgéneros descritos Amygdalus, Prunus, y Cerasus. Estas especies son denominadas como frutales de hueso y producen unos frutos llamados drupas con un gran valor comercial, incluyendo también el aprovechamiento de las semillas en algunas especies como el almendro (Figura 1). Un cuarto subgénero Eplectocladus ha sido descrito también aunque únicamente incluye especies silvestres de almendro sin uso comercial (Potter, 2012). Las especies de frutales de hueso cultivadas son originarias de Centro Asia y próximo oriente y están extendidas por todo el mundo con una superficie total en torno a los 34 millones de ha y una producción que excedió los 43 millones de toneladas (Mt) en 2013 (http://faostat.fao.org) concentrada sobre todo en Asia (19,5 Mt), Europa (16,5 Mt) y Norte América (5Mt) (Figura 1). Dentro de la producción mundial de estos frutales de hueso destaca la producción de 21,63 Mt de melocotonero y nectarina [P. persica (L.) Batsch]; 2,91 Mt de almendro [P. amygdalus (Batsch) syn. P. dulcis (Miller) Webb]; 11,52 Mt de ciruelo incluyendo ciruelo mirobolano (P. cerasifera Ehrh.), ciruelo europeo (P. domestica L.) y ciruelo japonés (P. salicina Lindl); 4,11 Mt de albaricoquero (P. armeniaca L.) y albaricoquero japonés (P. mume Sieb. et Zucc.); y 3,49 Mt de cerezo dulce (P. avium L.) y guindo (P. cerasus L.) (http://faostat.fao.org).
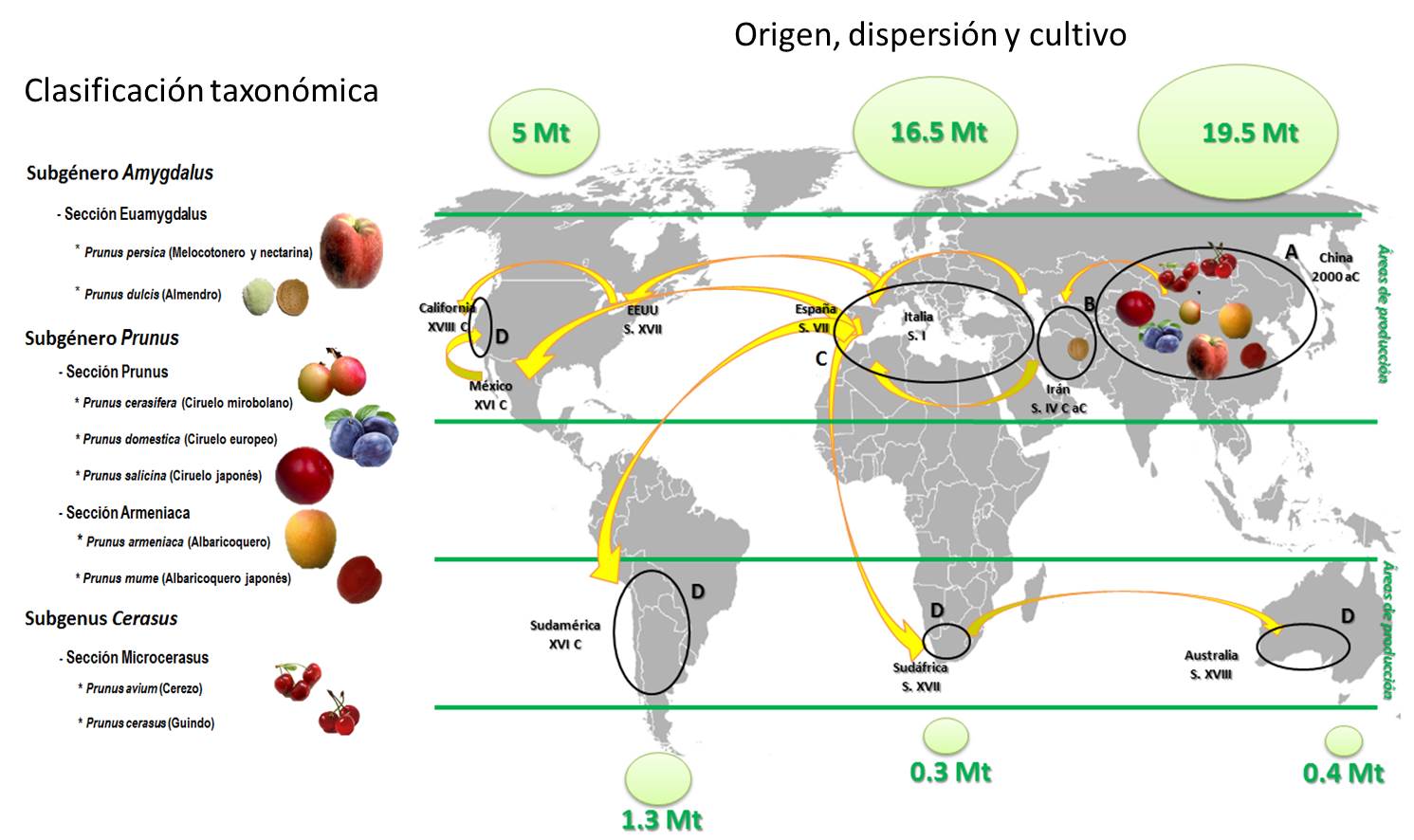
España con una producción de 1,8 Mt en 2013 se sitúa entre los principales productores a nivel mundial de estos frutales junto a EEUU, China e Italia. En 2013 España ocupó el tercer puesto a nivel mundial en producción de melocotón y nectarina con una producción de 1,32 Mt, el tercer lugar en la producción de almendra (149.000 t), décimo lugar en ciruela (172.000 t), octavo en albaricoque (131.000 t) y sexto en cereza (97.000 t). Esta producción se concentra principalmente en el arco mediterráneo (Cataluña, Valencia y Murcia) y Aragón, aunque también hay que señalar Extremadura como destacada productora de ciruela y sobre todo de cereza. Además, en nuestro país esta producción se dedica en gran medida a la producción de fruta para consumo en fresco y para la exportación a otros países, lo que da una idea del gran valor económico de esta producción. España es en estos momentos el principal país exportador de frutas de hueso. Los datos de exportación de estas frutas ofrecidos por la Federación Española de Asociaciones de Productores Exportadores de Frutas, Hortalizas, Flores y Plantas vivas (Fepex) en el año 2015 indican un volumen de exportación de 849.505 t (alrededor del 85% del total) de melocotonero y nectarina, 79.842 t de albaricoquero (60% del total), 108.716 t de ciruela (62%) y 26.371 t de cerezo (26%) (http://www.fepex.es). A las que hay que sumar las 65.000 t de almendro (30%) (http://www.magrama.gob.es). Estas cifras repercuten en un valor económico de estas exportaciones cifrado por Fepex en más de 1.100 millones de euros en 2015.
Además, en consonancia con la producción de calidad de estos frutales España es uno de los países junto a EEUU e Italia con un mayor número de programas de mejora de estas especies financiados con fondos públicos y un mayor dinamismo varietal. En estos momentos, existen siete centros de investigación públicos que desarrollan importantes programas de mejora específicos para las diferentes especies de frutales del género Prunus. Esta investigación, paralelamente a lo que ocurre con la producción, se concentra en el arco mediterráneo, Cataluña (los IRTA de Reus y Lleida), Valencia (el IVIA) y Murcia (el CEBAS-CSIC y el IMIDA), y Aragón (el CITA de Aragón y la estación de Aula Dei-CSIC). También hay que señalar el CICYTEX de Badajoz. En los centros de investigación del IRTA en Reus y Lleida se llevan a cabo programas de mejora de melocotonero y almendro. En Aragón, la Unidad de Investigación en Fruticultura del CITA de Zaragoza junto con la Estación Experimental de Aula Dei-CSIC están desarrollando varios programas de mejora genética de los frutales de hueso. Estos centros están especializados en la mejora genética de los patrones. El CITA además lleva a cabo programas de mejora de melocotonero y almendro y Aula Dei programas de melocotonero y albaricoquero. En Valencia los programas de mejora de melocotonero y albaricoquero se llevan a cabo en el IVIA. Los trabajos que se llevan a cabo en la Región de Murcia en el Departamento de Mejora Vegetal del CEBAS-CSIC y el Departamento de Fruticultura del IMIDA incluyen programas de mejora de las cinco grandes especies de Prunus, albaricoquero y almendro (CEBAS-CSIC), cerezo y melocotonero (IMIDA) y ciruelo entre ambas instituciones. Por último, en Extremadura el CICYTEX de Badajoz posee un programa de mejora genética de cerezo.
Análisis transcriptómico en frutales del género Prunus
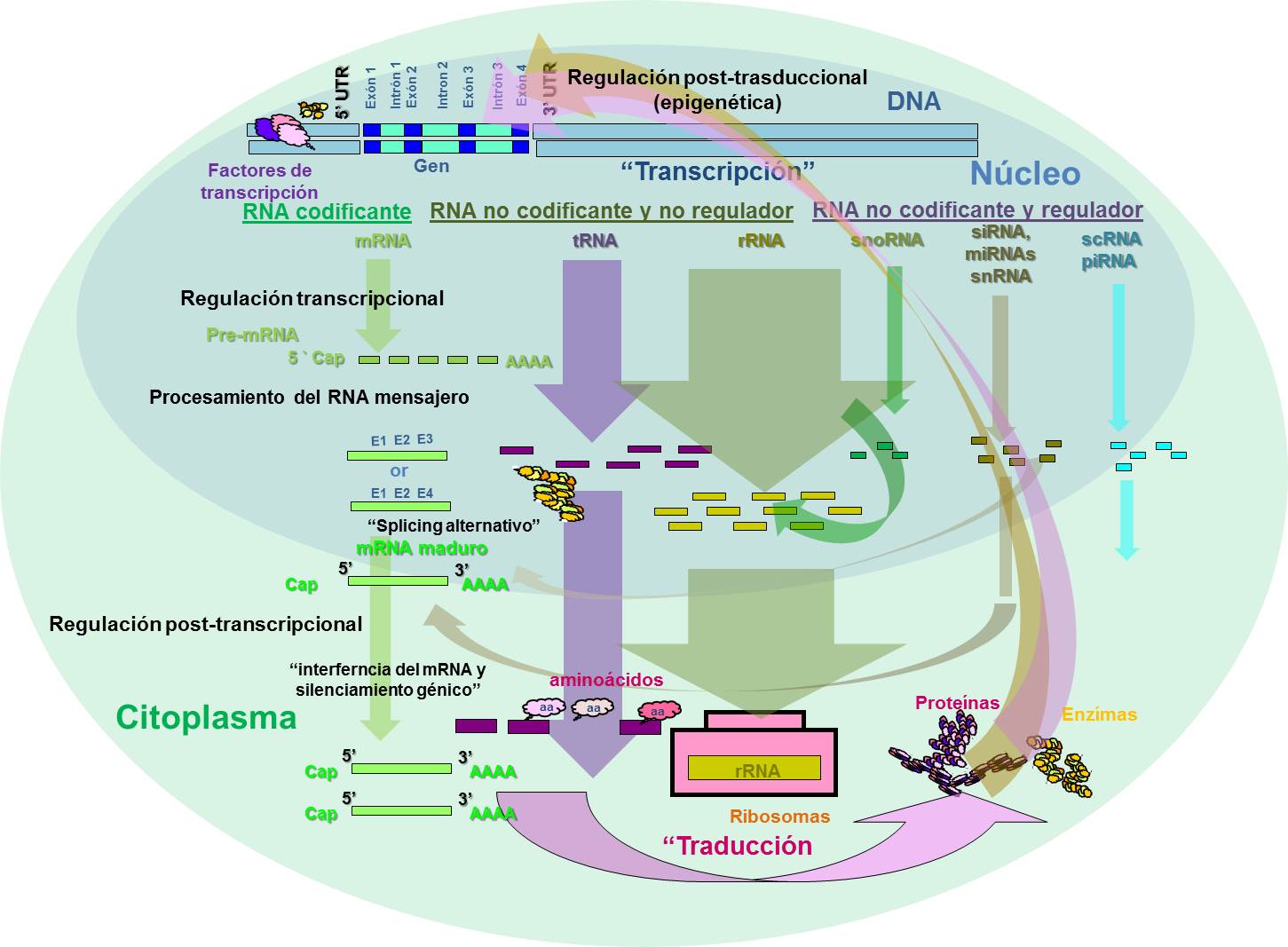
El transcriptoma (RNA) es una expresión flexible del genoma (DNA) capaz de ajustarse a las necesidades del desarrollo y de los requerimientos medioambientales de los organismos vivos. El transcriptoma está constituido físicamente por el RNA tanto codificante como no codificante. El RNA es una molécula mucho menos estable que el DNA lo que facilita poder ajustar su número de copias mediante reciclaje. El RNA que podemos encontrar en un organismo puede ser codificante como es el caso del llamado RNA mensajero (mRNA) que puede representar un 5% del total, pero mayoritariamente es RNA llamado no codificante. Este último RNA puede ser estructural como el de transferencia (tRNA) (5-10% del total) y el ribosómico (rRNA) ARNr que puede representar hasta un 85% del RNA total. A su vez podemos hablar dentro del RNA no codificante del RNA regulador (3-5% del total) que incluye siRNA (pequeño de interferencia) (20 nt), micro RNA (miRNA) (20-25 nt), snoRNA (de 60 a 300 nt) y otras clases de RNA como el small nuclear (snRNA), Piwi-interacting RNA (piRNA) o scRNA (Atkins et al., 2011; Watson, 2014) (Figura 2). El transcriptoma presenta una mayor diversidad que el genoma del que se origina ya que a partir de una misma región de DNA se pueden generar varias molecular de RNA y este RNA puede ser nuevamente procesado para volver a aumentar su diversidad.
El nudo gordiano del proceso de transcripción y expresión del DNA en un fenotipo concreto consiste en la traducción del mRNA maduro a la proteína; junto con la presencia de RNA no codificante y no regulatorio como el rRNA y tRNA; y la regulación post-transcripcional y post-trasduccional (epigenética) de este mRNA por parte de pequeños RNA no codificantes pero sí reguladores (miRNA, siRNA, piRNA, snRNA o snoARN) (Figura 2) (Martínez-Gómez et al., 2012). Las proteínas y el RNA forman una serie de redes que afectan a la expresión del ADN. Es esencial la comprensión del transcriptoma como molde para las proteínas y como elemento regulador para comprender los procesos de respuesta de la célula.
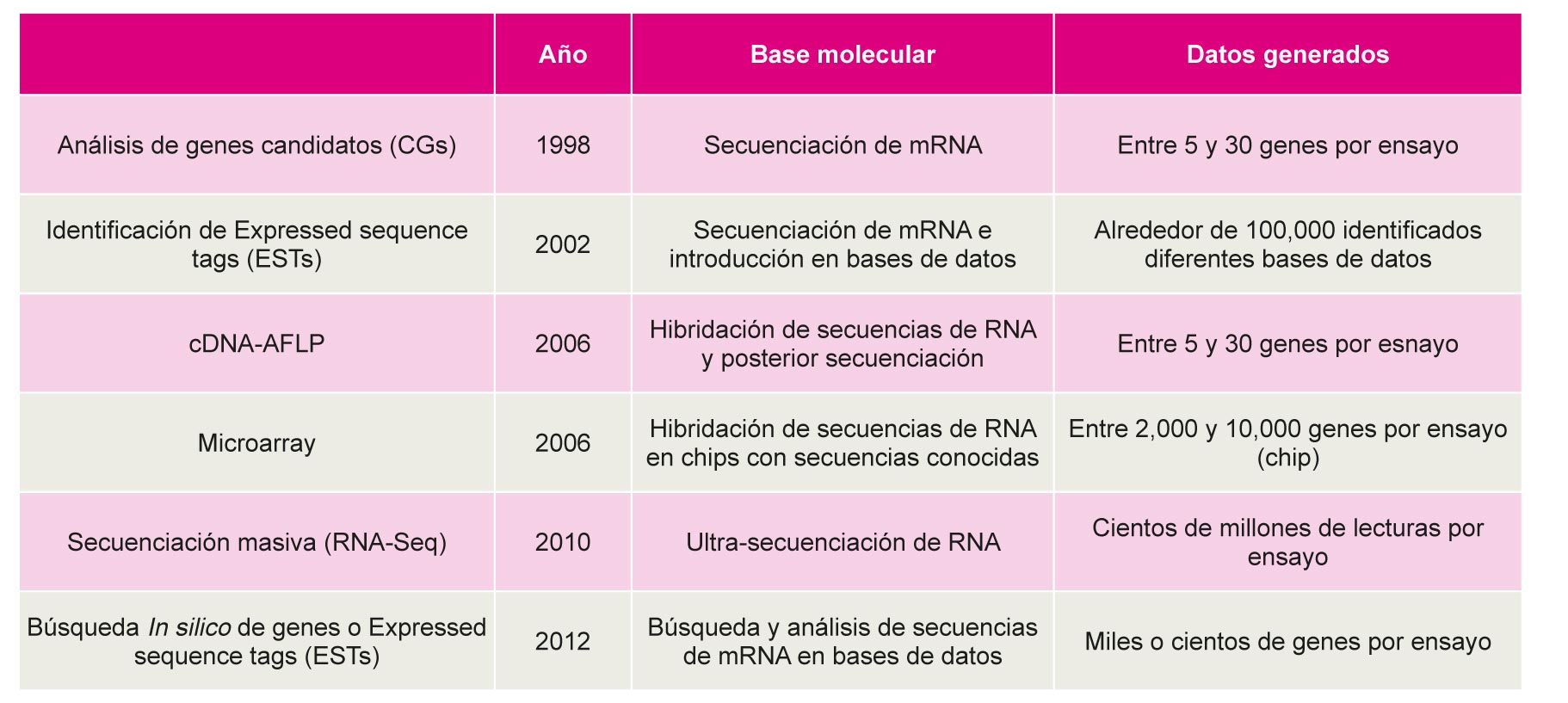
Los estudios iniciales en el transcriptoma de Prunus se orientaron al desarrollo y alineamiento de ESTs (Expressed sequence tags) y al análisis de los genes candidatos. El estudio de los ESTs en Prunus se inció en melocotonero, albaricoquero y almendro. Actualmente la base de datos del genoma Rosaceae (GDR) contiene más de 100.000 ESTs de distintas especies de Prunus (principalmente melocotonero). Otra aproximación posterior fue la de la hibridación de mRNAs mediante las técnica de cDNA-AFLP. Posteriormente se han desarrollado técnicas de análisis masivo de genes. La primera de las cuales fue el desarrollo del microarray, chips con unos miles de secuencias conocidas. Durante los últimos años, sin embargo, las llamadas metodologías de alto rendimiento ('high-throughput') para la secuenciación del DNA (DNA-Seq, en 2005) y cDNA proveniente del RNA (RNA-Seq, en 2008), están causando una revolución en la investigación biológica. Además, la tercera generación de estos ultra secuenciadores pretende ser capaz de determinar la composición de bases de ADN de una sola célula.
En estos momentos disponemos de información precisa sobre la constitución de DNA o RNA de una planta sin necesidad de un clonaje previo. Las nuevas tecnología de secuenciación están generando una enorme cantidad de datos cuyo manejo y almacenamiento son posibles mediante un desarrollo similar de los medios informáticos (Martínez-Gómez et al. 2011). Posteriormente los trabajos de análisis masivo han continuado mediante la denominada búsqueda in silico utilizando avanzadas herramientas bioinformáticas para analizar los datos de las bases de datos existentes. También se dispone de bases de datos con análisis de expresión cuantitativa de colecciones de ESTs de diferentes tejidos, genotipos y estados de desarrollo clasificados de acuerdo con el programa Gene Ontology (GO) (Cuadro 1).
En estos momentos podríamos pues hablar de una nueva 'era' en los estudios sobre la genética de los frutales del género Prunus (albaricoquero, almendro, melocotonero, ciruelo, etc.) y su aplicación en el desarrollo de nuevas variedades dentro de los distintos Programas de Mejora Genética. En este nuevo contexto (post-genómico) se presentan una serie de nuevos desafíos biológicos y oportunidades en la aplicación de toda la gama de las ciencias ómicas como la transcriptómica, en el desarrollo de estrategias eficaces de selección asistida por marcadores en Prunus. Estas oportunidades son de especial interés en el caso de Prunus, donde el conocimiento de la asociación entre genes y caracteres agronómicos es bastante limitado. En este contexto, se ha puesto en marcha un laboratorio de análisis transcriptómico de frutales de hueso dentro del Grupo de Mejora Genética de Frutales del CEBAS-CSIC de Murcia (Figura 3). Este laboratorio tiene como objetivo el análisis transcriptómico en frutales del género Prunus y el desarrollo de aplicaciones a los programas de mejora. Está dedicado a los trabajos de extracción del RNA para su posterior envío a los diferentes servicios de secuenciación masiva tanto de mRNA como de miRNA existentes en España y Europa; además del análisis bioinformático de los datos obtenidos mediante secuenciación y la validación de los datos obtenidos mediante RT-PCR cuantitativa (qPCR) o semicuantitativa (RT-PCR) ensayando los genes de interés uno a uno.

Aplicación de las técnicas de análisis transcriptómico en los programas de mejora de frutales
En el caso de especies del género Prunus, la secuenciación del transcriptoma (RNA-Seq) ha sido aplicada en melocotonero para facilitar y aislar genes que controlan diferentes caracteres agronómicos del árbol y el fruto (Wang et al., 2013; Chen et al., 2014; Sanhueza et al., 2015). También se ha utilizado para estudiar los genes implicados en la patogénesis de Xanthomonas arboricola (Socquet-Juglard et al., 2013) o Lasiodiplodia theobromae (Gao et al., 2016) en melocotonero. En otras especies, esta tecnología ha sido utilizada en el análisis de expresión génica de la dormancia de la yema en albaricoquero japonés (Zhong et al., 2013), el desarrollo del fruto (Alkio et al., 2014) y calidad del fruto (Wei et al., 2015) en cerezo dulce, o la resistencia al frío en almendro (Mousavi et al., 2014). Un ejemplo reciente de aplicación de esta técnica de RNA-Seq para el mejor conocimiento de los procesos biológicos y moleculares asociados a la expresión de los caracteres de interés agronómico es la expresión de la susceptibilidad/resistencia a Plum pox virus (PPV, sharka) en frutales del género Prunus (Rubio et al. 2015a; 2015b).
Estos estudios pusieron de manifiesto que la respuesta temprana a la infección por PPV en melocotonero y albaricoquero está asociada a una inducción de genes relacionados con la resistencia a patógenos como ácido jasmónico, proteínas de resistencia, quitinasas, citoquininas o proteínas Lys-M. En cambio, cuando el virus se había establecido, se observaba una sobreexpresión de genes Dicer protein 2a que podían evidenciar también una respuesta de silenciamiento génico de la planta suprimida mediante las proteínas HCPro y P1 de PPV. Este fenómeno de silenciamiento génico puede ser la causa de la eliminación de síntomas del virus en plantas susceptibles ya infectadas. Por otro lado, acerca de la resistencia a PPV en albaricoquero, los resultados obtenidos indican que genes tipo RTM [Restricted Tobaco etch virus (TEV) movement] dentro de la familia de genes de homología de dominio MATH parecen ser los principales implicados en el control de esta resistencia. Además de estos genes otros genes candidatos implicados en la resistencia son Pleiotropic drug resistance 9 gene; Cysteine-rich secretory proteins, Antigen 5 and Pathogenesis-related 1 protein (CAP) y Late embryogenesis abundant protein (LAE) (Figura 4).
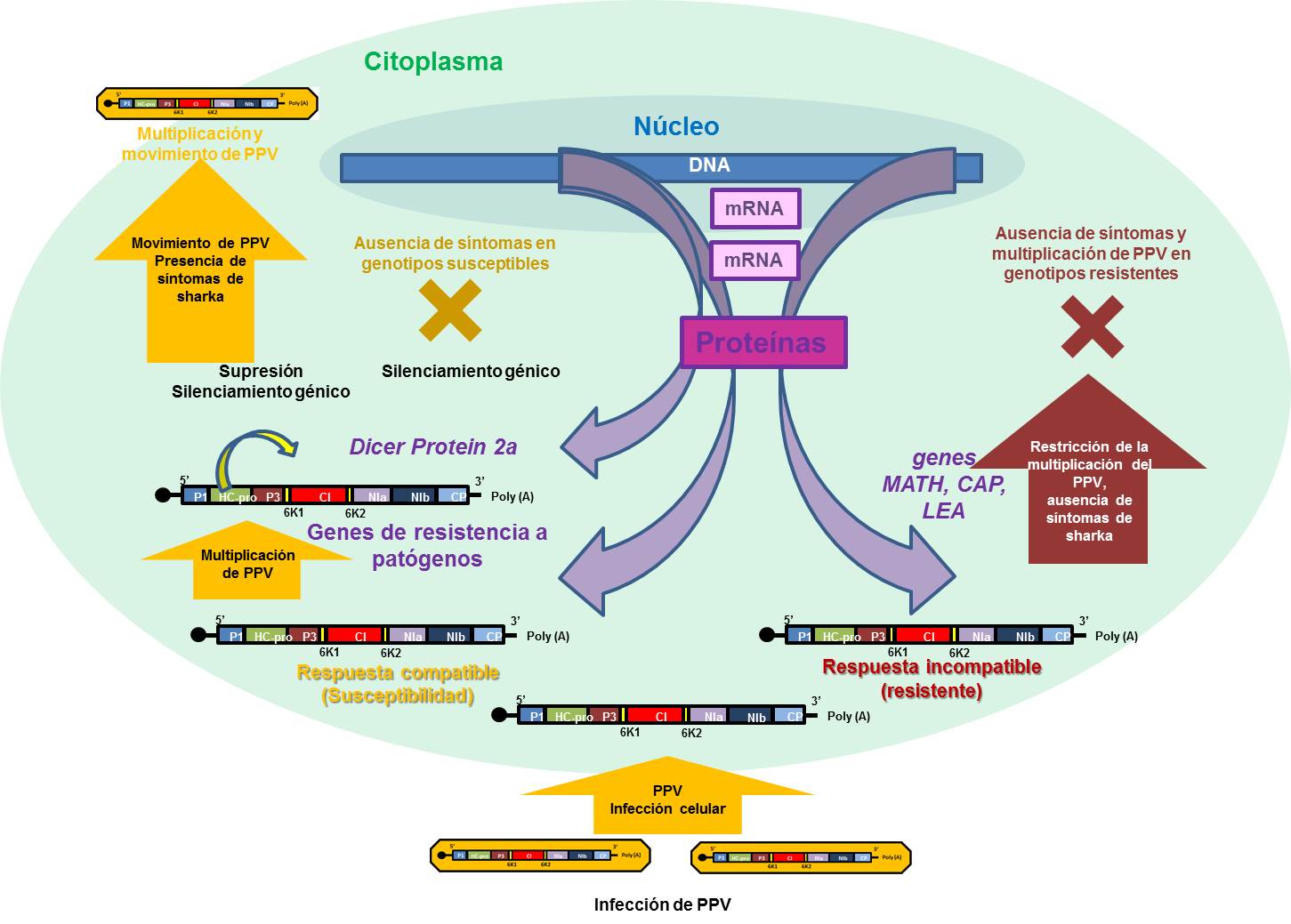
Figura 4. Representación esquemática de la interacción entre PPV (Plum pox virus) y frutales del género Prunus estudiada mediante la secuenciación masiva de diferentes transcriptomas de genotipos de frutales (albaricoquero y melocotonero) susceptibles y resistentes. Adaptación de los trabajos de Rubio et al. (2015a y 2015b).
El RNA-Seq está sirviendo también para identificar SNPs en estas regiones exómicas y en muchos casos en genes que han sido anotados en el genoma de referencia del melocotonero que como hemos comentado en el futuro se pueden asociar a caracteres agronómicos de interés mediante análisis de QTLs (Quantitave trait loci) y MTLs (Mendelian trait loci) (Salazar et al. 2014). Entre las especies secuenciadas se encuentra el melocotonero la cual sirve de genoma de referencia de los estudios genéticos donde comparamos la expresión diferencial entre una situación normal y otra atípica, como una enfermedad, y de este modo podemos conocer los genes que participan en el proceso de defensa, por ejemplo. En Prunus, se estima la existencia de 27.852 genes que son responsables de las distintas características. La secuenciación del genoma completo del melocotón representa el principal hito de la era de la genómica en especies de Prunus. En 2010 se publicó en la red la primera secuencia completa de un genoma de Prunus, proveniente de un genotipo de melocotonero (www.rosaceae.org) (Verde et al., 2013). Esta información permite localizar en estos genomas de referencia los genes expresados, lo que puede ser más importante que los QTLs y MTLs desarrollados mediante la genética clásica de ligamiento que son en estos momentos la fuente más extensa de información sobre la genómica funcional de estas especies arbóreas.
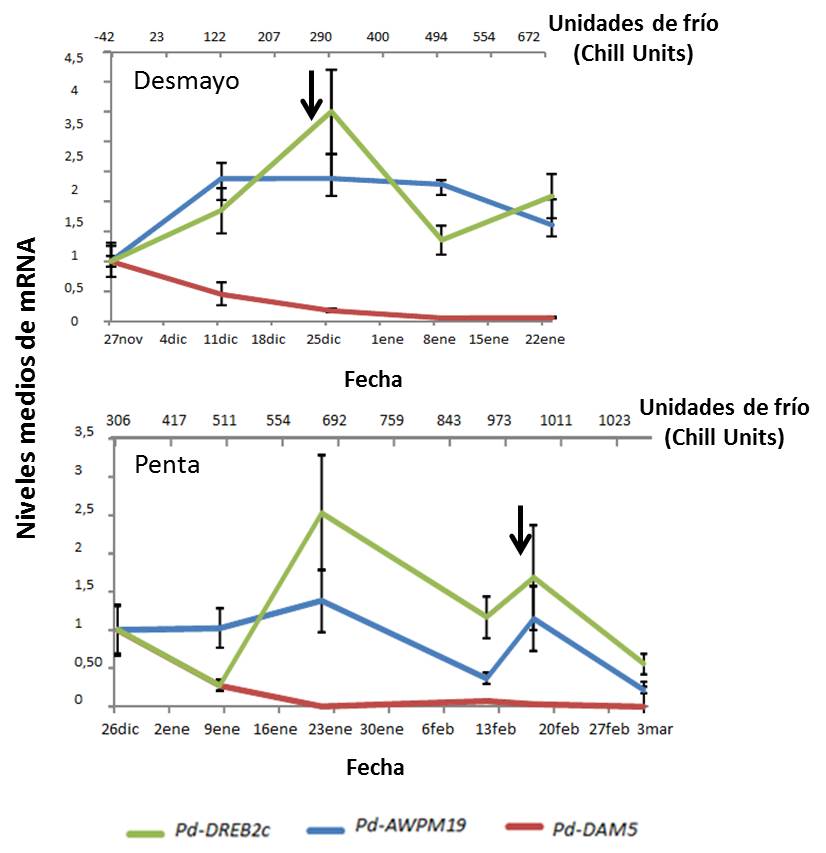
Finalmente, como marcadores para selección asistida estas técnicas de análisis del RNA están en proceso de desarrollo con un gran potencial pero con una aplicación actual muy reducida en comparación con los marcadores de DNA. Un ejemplo reciente de aplicación de técnicas de qPCR para el análisis de la expresión de un carácter como la época de floración en almendro ha sido presentado por Prudencio et al. (2016). El almendro florece como respuesta a una pauta establecida de bajas y altas temperaturas después de la ruptura del letargo invernal. Estas necesidades de frío y calor garantizan que en cada zona la floración tendrá lugar en un momento favorable para la polinización. Prudencio et al. (2016) han ensayado en yemas de flor la expresión de varios genes en dos variedades de almendro, una de floración tardía (‘Penta’) y otra de floración temprana (‘Desmayo Largueta’), durante 10 fechas diferentes antes y después de la salida del letargo. Después del letargo de las yemas la inducción en la expresión de algunos genes se reduce drásticamente. Estos resultados ponen de manifiesto el papel de estos genes en el mantenimiento de la dormancia y el uso potencial de un marcador mediante la aplicación de la qPCR para detectar la salida del letargo en la especie almendro (Figura 5).
Agradecimientos
Este trabajo ha sido financiado por los proyectos 'Regulación de la expresión génica en la resistencia al Plum pox virus (Sharka) inducida en melocotonero mediante el injerto del almendro Garrigues' (AGL2013-43550-R) del Ministerio de Economía y Competitividad, y el proyecto 'Breeding stone fruit species assisted by molecular tools' (19879/GERM/15), financiado dentro del Programa de Ayudas a los Grupos y Unidades de Excelencia Científica de la Región de Murcia de la Fundación Séneca de Murcia.
Referencias bibliográficas
- Alkio, M., Jonas, U., Declerq, M., Van Nocker, S., Knoche, M. 2014. Trancriptional dynamics of the developing sweet cherry (Prunus avium L.) fruit: sequencing, annotation and expression profiling of exocarp-associated genes. Horticulture Research 1: 11
- Atkins, J.F., Gesteland, R.F., Cech, T.R. 2011. RNA Worlds: From life's to diversity in gene regulation. Cold Spring Harbor: Cold Spring Harbor Laboratory Press, New York, USA. 361 pp.
- Chen, Y., Mao, Y., Liu, H., Yu, F., Li, S., and Yin, T. 2014. Transcriptome differentially expressed genes relevant to variegation in peach flowers. PLOS ONE 9: e90842
- Gao, L., Wang, Y.T., Li, Z., Zhang, Z., Ye, J.L., Li, G.H.. 2016. Gene Expression Changes during the Gummosis Development of Peach Shoots in Response to Lasiodiplodia theobromae Infection Using using RNA-Seq. Frontiers in Physiology 7: 170
- Martínez-Gómez, P., Crisosoto, C., Bonghi, C., Rubio, M. (2011) New approaches to Prunus transcriptome analysis. Genetica 139: 755–769.
- Martínez-Gómez, P., Sánchez-Pérez, R., Rubio, M. 2012. Clarifying omics concepts, challenges and opportunities for Prunus breeding in the post-genomic era. OMICS: J. Integrative Biology 16: 268–283.
- Mousavi, S., Alisoltani, A., Shiran, B., Fallahi, H., Imani, A., Houshmand, S. 2014. De Novo Transcriptome Assembly and Comparative Analysis of Differentially Expressed Genes in Prunus dulcis Mill. in Response to Freezing Stress. PLOS One 10: e104541
- Potter D. 2012. Basic information on the stone fruit crops. In Genetics, Genomics and Breeding of Stone Fruits. C. Kole, and A.G. Abbott eds. CRC Press, New York, USA. pp: 1–21
- Prudencio AS, Del Cueto J, Dicenta F, Sánchez-Pérez R, Martínez-Gómez P. 2016. Análisis de la expresión génica durante la salida del letargo de las yemas de flor en almendro. X Jornadas de Fruticultura-SECH (FrutiSech2016). Enero de 2016. Murcia
- Rubio, M., Rodríguez-Moreno, L., Ballester, A.R., Castro de Moura, M., Bonghi, C., Candresse, T., Martínez-Gómez, P. 2015a. Analysis of gene expression changes in peach leaves in response to Plum pox virus infection using RNA-Seq. Molecular Plant Pathology 16: 164-176
- Rubio, M., Ballester, A.R., Olivares, P.M., Castro de Moura, M., Dicenta, F., Martínez-Gómez, P. 2015b. Gene expression analysis of Plum pox virus (Sharka) Susceptibility/Resistance in apricot (Prunus armeniaca L.). PLOS One 10: e0144670
- Salazar, J.A., Ruiz, D., Campoy, J.A. et al. 2014 Quantitative Trait Loci (QTL) and Mendelian Trait Loci (MTL) analysis in Prunus: A breeding perspective and beyond. Plant Molecular Biology Reporter 32: 1-18
- Sanhueza, D., Vizoso, P., Balic, I., Campos-Vargas, R., Meneses, C. 2015. Transcriptomic analysis of fruit stored under cold conditions using controlled atmosphere in Prunus persica cv. 'Red Pearl'. Frontiers in Plant Science 6: 788
- Socquet-Juglard, D., Kamber, T., Pothier, J.F., Christen, D., Gessler, C., Duffy, B., Patochi, A. 2013. Comparative RNA-Seq analysis of early-infected peach leaves by the Invasive phytopathogen Xanthomonas arboricola pv. pruni. PLoS ONE 8: e54196
- Verde, I., Abbott, A.G., Scalabrin, S., et al. 2013. The high-quality draft of peach (Prunus persica) identifies unique patterns of genetic diversity, domestication and genome evolution. Nature Genetics 45: 487–494.
- Wang, L., Zhao, S., Gu, C., Zhou, Y., Zhou, H., Ma, J., Cheng, J, Han, Y. 2013. Deep RNA-Seq uncovers the peach transcriptome landscape. Plant Mol. Biology 83: 365–377.
- Watson, J.D., Gann, A., Baker, T.A., Levine, M., Bell, S.P., Harrison, S.C. 2014. Molecular Biology of the gene. Cold Spring Harbor: Cold Spring Harbor Laboratory Press, New York, USA. 872 pp.
- Wei, H.R., Chen, X., Zong, X.J., Shu, H.R., Gao, D.S., Li, Q.Z. 2015. Comparative Transcriptome Analysis of Genes Involved in Anthocyanin Biosynthesis in the Red and Yellow Fruits of Sweet Cherry (Prunus avium L.). PLOS One 10: e0121164
- Zhong, W., Gao, Z., Zhuang, W., Shi, T., Zhang, Z., Ni, Z. 2013. Genome-wide expression profiles of seasonal bud dormancy at four critical stages in Japanese apricot. Plant Mol. Biology 86: 247–264