Detectando todos los potyvirus que infectan un cultivo en un único test
Jesús A. Sánchez-Navarro1, Christopher N. Cooper2 y Vicente Pallás1
1 Departamento de Virología Molecular y Evolutiva de Plantas. Instituto de Biología Molecular y Celular de Plantas (IBMCP) (UPV-CSIC)
2 Georgia Institute of Technology. School of Chemistry & Biochemistry, Atlanta. USA
20/05/2019Durante muchos años el principal objetivo de los estudios en el campo del fitodiagnóstico ha tratado de ir superando umbrales de sensibilidad que han permitido detectar a día de hoy a nivel de molécula. Los retos en esta disciplina se han desplazado recientemente a la consecución de formatos multidiana. En este sentido, el uso de ‘polisondas’, o sondas de RNA que llevan secuencias parciales de diferentes virus o viroides de plantas fusionados en tándem, ha permitido la detección polivalente de hasta diez patógenos diferentes mediante un procedimiento basado en hibridación molecular no radiactiva. En el presente trabajo, hemos desarrollado una nueva polisonda con la capacidad de detectar a todos los miembros del género Potyvirus, que hemos denominado 'sonda de género'. Para ello, hemos explotado la capacidad del ensayo de hibridación molecular para hibridar de forma cruzada con secuencias que muestran un porcentaje de similitud del 68% o más, al reducir la temperatura de hibridación. De acuerdo con esto, hemos desarrollado una polisonda que fue capaz de detectar los 32 potyvirus analizados en el presente trabajo sin señal en el tejido sano.

Los virus de las plantas siguen siendo uno de los principales contribuyentes a las pérdidas económicas en la agricultura. Se ha estimado que los virus de plantas pueden causar hasta 50 mil millones de euros en pérdidas anuales en todo el mundo. Este escenario puede agravarse debido esencialmente a los efectos que el cambio climático está teniendo sobre los vectores de estos patógenos y sus correspondientes huéspedes. Los métodos de detección temprana siguen siendo una de las principales y más efectivas estrategias de control para las enfermedades virales de las plantas. Durante los últimos años, se ha avanzado considerablemente en el desarrollo de herramientas con alta especificidad y bajos límites de detección para su uso en la detección de estos patógenos de plantas. Las reducciones de tiempo y costes han sido algunos de los principales objetivos perseguidos durante los últimos años, ya que aumentan su viabilidad para el uso rutinario.
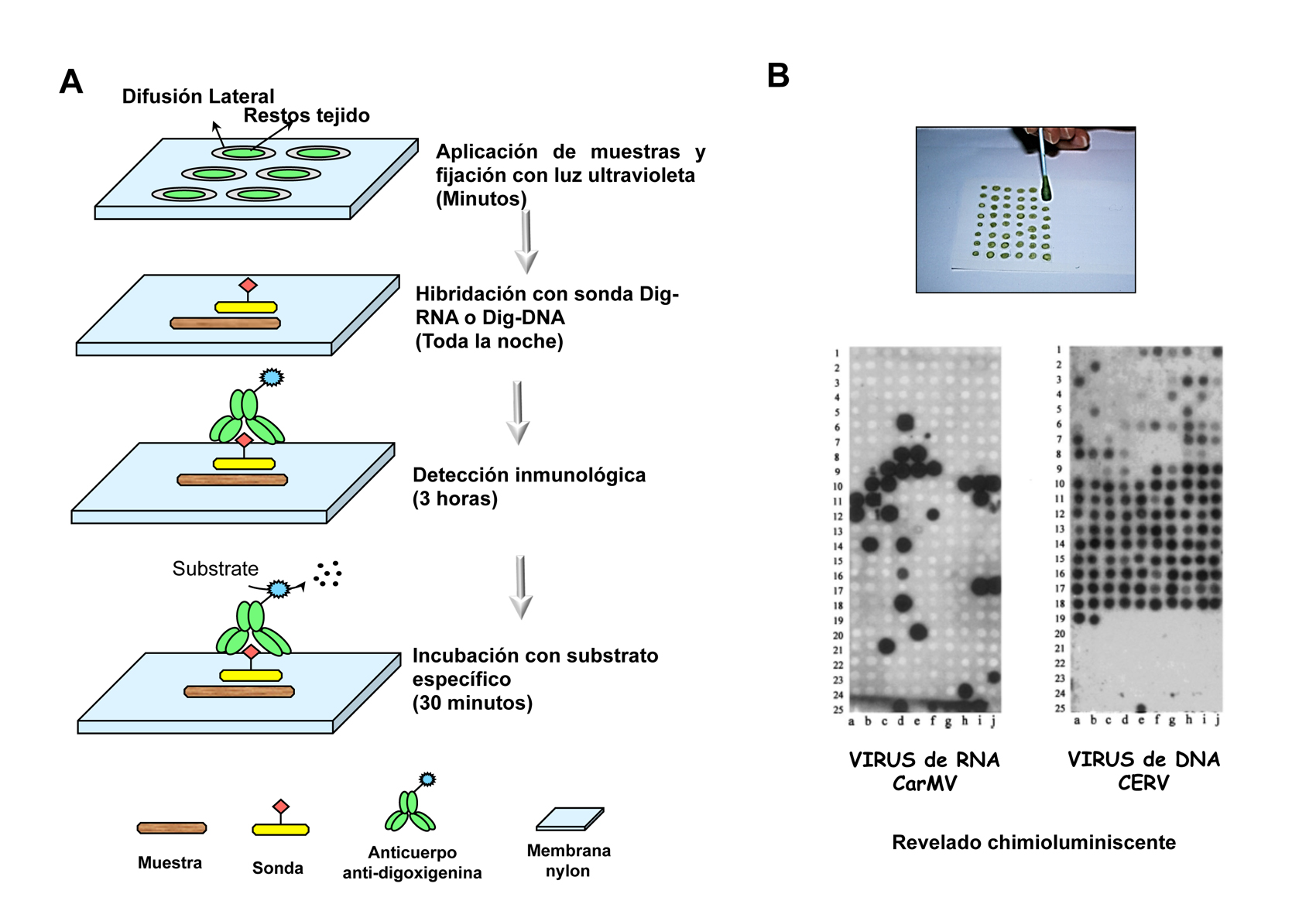
Los métodos de detección tradicionales incluyen bioensayos con plantas indicadoras y métodos serológicos como el ensayo immunoabsorbente ligado a enzimas (ELISA). En las últimas décadas se han incorporado las técnicas de detección molecular (RT-PCR, microarrays, hibridación molecular, etc.) en el diagnóstico de rutina de los virus de plantas (Jeong et al. 2014; Lopez et al. 2003). La detección múltiple mediante el uso de la técnica de hibridación molecular no isotópica (Figura 1) se abordó en un principio mediante un cóctel de las sondas individuales específicas en la solución de hibridación, que permite la detección polivalente de los diferentes virus que afectan a plantas ornamentales (Sánchez-Navarro et al. 1999), hortícolas (Minutillo et al. 2012; Saldarelli et al. 1996) y frutales de hueso (Saade et al. 2000). Posteriormente, nuestro grupo desarrolló la tecnología basada en las ‘polisondas’ o sondas de RNA que contienen secuencias parciales de ácido nucleico de diferentes virus (Aparicio et al. 2009; Herranz et al. 2005; Peiro et al. 2012) o viroides (Cohen et al. 2006; Lin et al. 2011; Zhang et al. 2012) clonados en tándem. Las polisondas han permitido la detección simultánea de seis virus diferentes (Herranz et al. 2005) o hasta ocho viroides (Cohen et al. 2006; Torchetti et al. 2012), ocho virus más dos viroides (Peiro et al. 2012) o tres patógenos diferentes que comprenden virus, viroides y bacterias (Zamora-Macorra et al. 2015), con el mismo límite de detección que las sondas individuales.
Potyvirus es un género de virus de la familia Potyviridae y representa uno de los grupos de virus vegetales más importantes desde el punto de vista económico y más ampliamente distribuidos. Los miembros de este género pueden producir pérdidas significativas en cultivos agrícolas, hortícolas y ornamentales. El miembro tipo del género Potyvirus, virus Y de la patata (PVY), junto con el virus A de la patata (PVA; género Potyvirus) y el virus del enrollamiento de la hoja de la patata (género Polerovirus), representa la máxima amenaza para la producción de patatas en todo el mundo y puede reducir los rendimientos de los cultivos hasta un 90% (Figura 2). El virus de la sharka (PPV; género Potyvirus) también tiene una gran importancia económica ya que ocasiona la enfermedad más destructiva de los frutales de hueso en todo el mundo. No es de extrañar, por tanto, que el PVY y el PPV se hayan seleccionado entre los diez virus vegetales de mayor importancia económica y científica (Scholthof et al. 2011). El hecho de que los potyvirus se propaguen por áfidos, y de que cada potyvirus pueda transmitirse por muchas especies de áfidos diferentes y de que cada especie de áfido pueda transmitir muchos potyvirus, hace incluso más difícil combatir y prevenir las infecciones por potyvirus en la agricultura.

En el presente trabajo, hemos explorado la capacidad potencial de la tecnología de polisondas para desarrollar una sonda universal con la propiedad de detectar a todos los miembros de un género viral específico, en este caso el género Potyvirus. La hibridación cruzada observada a 50-55°C entre secuencias que comparten una identidad del 68% o más, se aprovechó para diseñar varias polisondas, que llevan en tándem diferentes fragmentos de 500 nucleótidos de la región conservada del gen NIb de los potyvirus, con la capacidad para hibridar, de forma cruzada, con todas las especies de potyvirus disponibles en las bases de datos. Una polisonda que lleva 7 regiones conservadas diferentes fue capaz de detectar los 32 potyvirus analizados en el presente trabajo sin observarse ninguna respuesta cruzada en el tejido sano, lo que indica la capacidad potencial de estas sondas para detectar todos los potyvirus descritos, y probablemente los no caracterizados todavía.
Resultados y Discusión
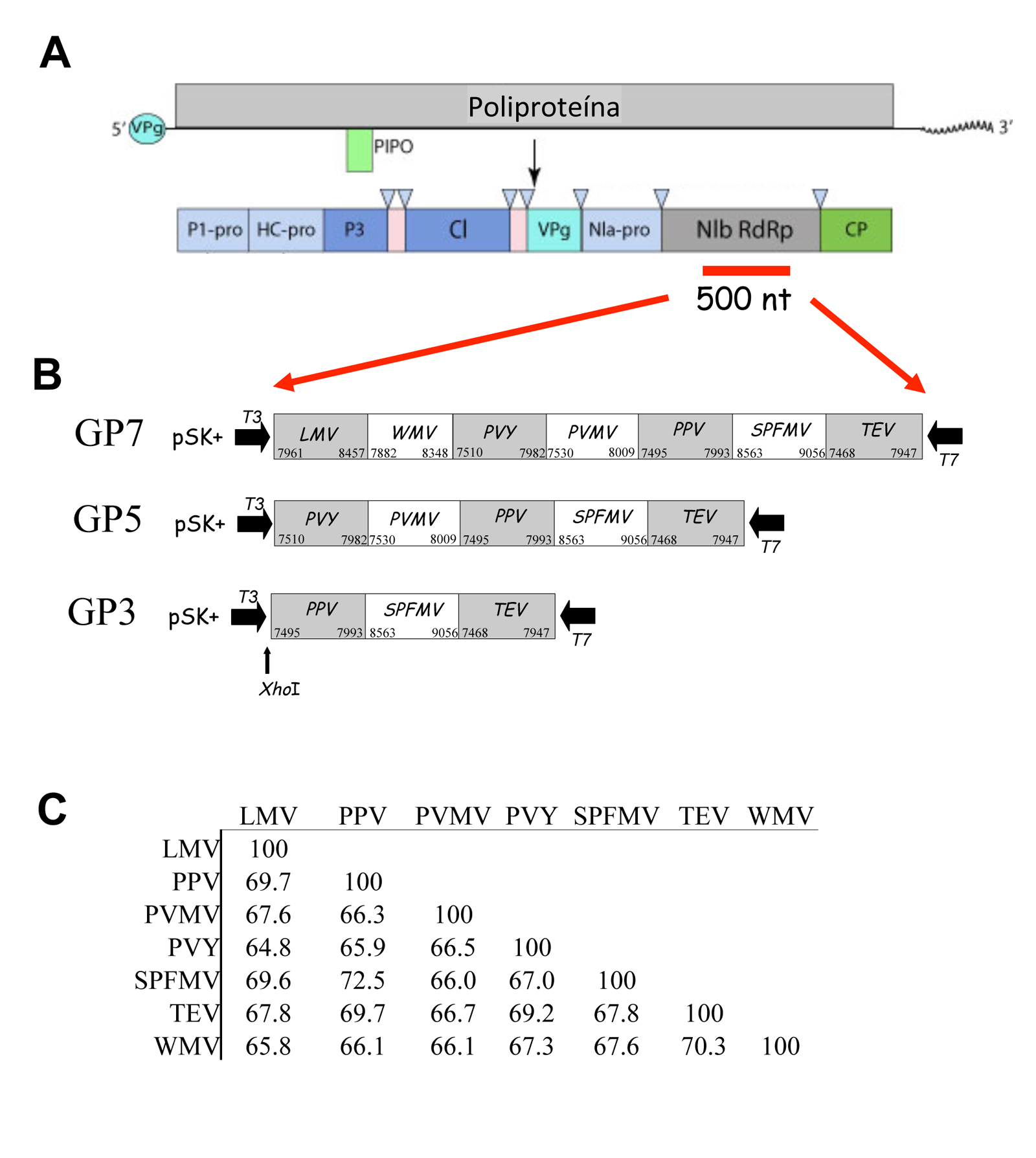
Previo al diseño de polisonda de género, realizamos un alineamiento con todas las secuencias completas de potyvirus (94 secuencias) disponibles en las bases de datos, utilizando para ello el programa Mega X (Kumar et al. 2018). Los resultados obtenidos pusieron de manifiesto que la región más conservada entre todas las secuencias correspondió a un fragmento de unos 500 residuos de nucleótidos localizadas en el gen de inclusión nuclear b (NIb) (Figura 3A). Para la síntesis de polisondas de género, seleccionamos siete regiones altamente conservadas distribuidas por el árbol filogenético de potyvirus y correspondientes a los virus: virus del mosaico de la lechuga (LMV), virus del mosaico de la sandía (WMV), virus Y de la patata (PVY), virus del moteado de las venas del pimiento (PVMV), virus de la sharka (PPV), virus del moteado suave de la patata (SPFMV) y virus del grabado del tabaco (TEV) (Figura 3B). Todas las secuencias seleccionadas presentaron porcentajes de identidad que oscilaron entre 64,8 y 72,5 (Figura 3C). Los fragmentos seleccionados se clonaron en el vector plasmídico pSK+ que posee los promotores de las RNA polimerasas T7 y T3, permitiendo la síntesis de transcritos de ambas polaridades del fragmento introducido (Figura 3B). Las siete regiones seleccionadas se clonaron individualmente o fusionadas en tándem para generar tanto las sondas individuales como las polisondas de género con 3 (GP3), 5 (GP5) o los 7 fragmentos (GP7), respectivamente.
Una vez generadas las diferentes sondas marcadas con digoxigenina (Roche) (Pallas et al. 1998), analizamos su capacidad para hibridar de forma cruzada con secuencias con altos porcentajes de identidad. Para ello, se sintetizaron transcritos complementarios a las 7 regiones seleccionadas y se prepararon réplicas de la misma membrana que contenía cantidades conocidas de cada transcrito (Figura 4). La hibridación de esta membrana con la sonda específica de PPV a diferentes temperaturas puso de manifiesto que no hay hibridación cruzada cuando el ensayo se realzó a 68 °C. Sin embargo, la reducción de la temperatura de hibridación de 8 grados (60 °C) fue suficiente para observar hibridación cruzada con el fragmento de TEV, a una concentración de 200pg/µl, mientras que la reducción de 5 (55 °C) o 10 (50 °C) grados fue suficiente para detectar dos (TEV, SPFMV) o todas las secuencias a diferentes concentraciones (Figura 4A), respectivamente. En la hibridación realizada a 50 °C se observó una señal suave con RNA total extraído de tejido sano indicando que a esta temperatura las sondas individuales reconocen inespecíficamente otros RNA celulares o con un porcentaje de identidad menor. El resto de sondas individuales se ensayaron con réplicas de la misma membrana y a diferentes temperaturas observando que todas dieron hibridación cruzada con secuencias que presentaban un porcentaje de identidad superior al 67%, mientras que las secuencias que tenían un porcentaje de identidad entre el 65% y 67% dieron un comportamiento diferente indicando que otros factores, aparte de la identidad, pueden influir en la hibridación cruzada (Figura 4B).
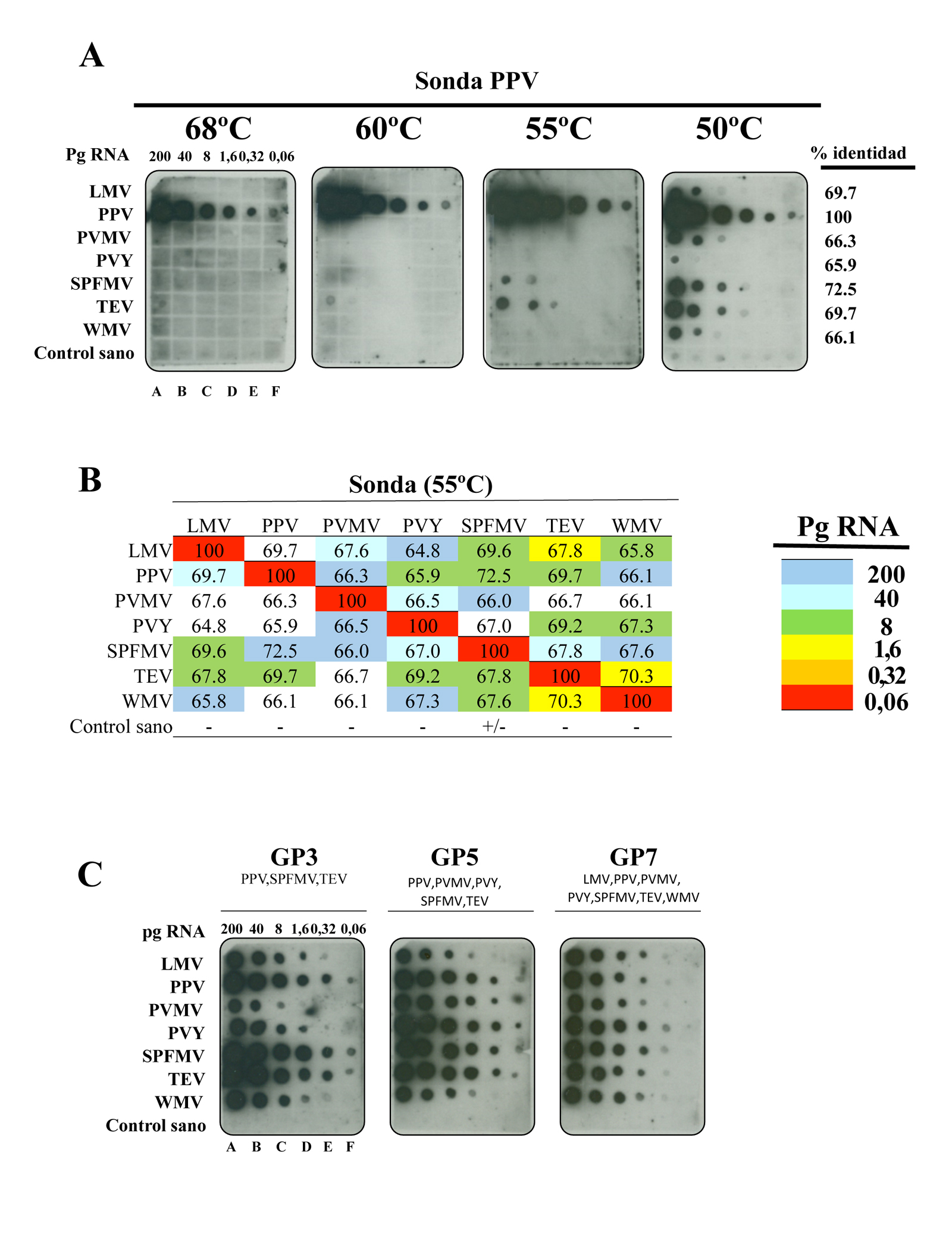
En el siguiente paso evaluamos las tres sondas de género que habíamos generado (GP3, GFP5 y GP7) utilizando réplicas de la misma membrana ensayada con las sondas individuales, con la salvedad de que las hibridaciones se realizaron a 50 °C al no observar hibridación cruzada con RNA total de tejido sano (Figura 4C). Los resultados obtenidos pusieron de manifiesto que las tres polisondas de género detectaron todos los virus hasta una concentración de 8 pg/µl (GP3), 1.6 pg/µl (GP5) o 0.32 pg/µl (GP7), sin obtener señal en RNA total extraído de tejido sano de diferentes huéspedes. Estos resultados sugerían que las diferentes polisondas de género podrían hibridar de forma cruzada con cualquier secuencia que presentara un porcentaje de identidad del 67% o superior a las secuencias presentes en cada polisonda.
Para predecir los posibles virus que podrían ser detectados realizamos un análisis in silico comparando todas las secuencias de potyvirus depositadas en la base de datos con los 7 fragmentos incluidos en las diferentes GP mediante el programa MatGAT (Campanella et al. 2003). Los resultados obtenidos indicaban que todos los potyvirus presentes en la base de datos presentaban un porcentaje de identidad del 68% o superior con al menos dos de los siete fragmentos seleccionados para hacer las GP. Solo el virus del enanismo amarillo de la cebolla (Onion yellow dwarf virus, OYDV) presentó un porcentaje de identidad del 69,3% solo con el fragmento de PPV.
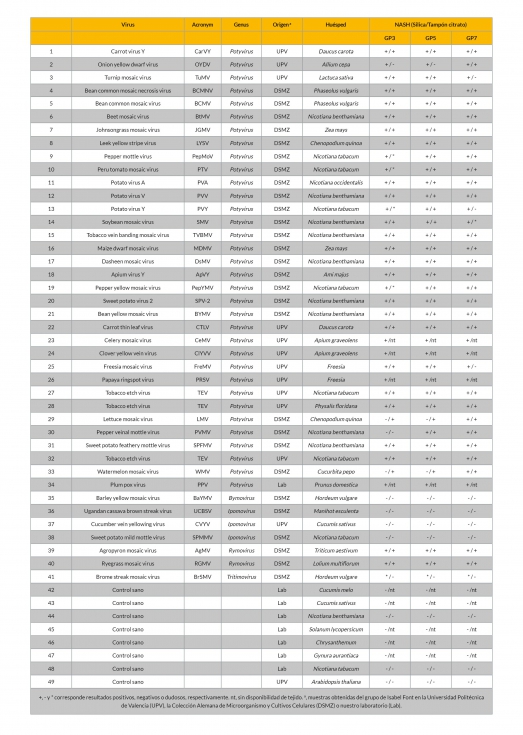
En este estudio también incluimos otras especies de la familia Potyviridae observando que a excepción del género Rymovirus, el resto de virus presentaron un porcentaje de identidad inferior al 65%. En el caso de rymovirus, las tres especies incluidas presentaron porcentajes de identidad del 68%, indicando que podrían ser detectadas por la GP de potyvirus. En conjunto, los resultados obtenidos indicaban que todos los potyvirus presentes en la base de datos podrían ser detectados con alguna de las tres GP desarrollados en el presente trabajo. Para confirmar esta hipótesis decidimos analizar 49 muestras vegetales de 7 especies diferentes, representativas de infecciones individuales de 32 potyvirus diferentes, 1 bymovirus, 3 ipomovirus, 2 rymovirus y 1 tritimovirus (Tabla 1). Las muestras se obtuvieron de la Colección Alemana de Microrganismos y Cultivos Celulares (German Collection of Microorganisms and Cell Cultures, DSMZ) y del Instituto Mediterráneo Agroforestal de la Universidad Politécnica de Valencia. El análisis de las muestras vegetales se realizó bien a partir de ácidos nucleicos totales (NASH) extraídos mediante el método de Silica (MacKenzie et al. 1997) o bien con un protocolo rápido homogenizando el tejido con tampón citrato sódico y aplicando directamente en la membrana (Sánchez-Navarro et al. 1998). Los resultados obtenidos pusieron de manifiesto que las 32 especies de potyvirus analizadas, así como los 2 rymovirus, fueron detectados mediante el análisis de NASH con las tres GP, a excepción de PVMV que no se detectó con GP3. En este sentido y a diferencia de GP5 y GP7, hay que destacar que el porcentaje de identidad de PVMV con los tres fragmentos incluidos en GP3, estaba el límite para poder ser detectado por hibridación cruzada (66-68%). Cuando el tejido infectado se analizó utilizando el protocolo rápido, se detectaron el 90,2% de los positivos detectados por NASH.
Los resultados obtenidos en el presente trabajo han puesto de manifiesto que el uso de sondas de RNA marcadas con digoxigenina y dirigidas a una región de 500 nt del gen NIb de potyvirus, permite la detección cruzada de diferentes especies cuando el porcentaje de identidad es superior al 68%. La utilización de polisondas con diferentes fragmentos fusionados en tándem que cubren la diversidad observada en esta región dentro del género Potyvirus, ha permitido detectar todas las especies ensayadas (32) junto con 2 rymovirus, indicando la capacidad potencial de la GP de detectar todos los potyvirus descritos y los no caracterizados. Además, la posibilidad de usar un protocolo rápido, que detectó el 90% de las muestras positivas analizadas por NASH, junto con la solidez de la metodología, que permite la detección de especies con una variabilidad significativa de secuencias de nucleótidos o incluso potyvirus no caracterizados, hace que la hibridación no radioactiva utilizando una GP sea una alternativa muy apropiada para el diagnóstico de rutina de potyvirus en grandes prospecciones. Actualmente, el uso del anticuerpo monoclonal MAb PTY1 de amplio espectro (Jordan and Hammond 1991), combinado con un procedimiento directo de extracción de tejido, representa una herramienta valiosa para las aplicaciones de diagnóstico y detección de potyvirus transmisibles por áfidos, aunque no se conoce cómo de amplio es el espectro de potyvirus que se podría detectar el anticuerpo MAb PTY1. Las sondas de género desarrolladas en este trabajo anticipan que todos los potyvirus pueden ser detectados (porcentaje de identidad de 68/69% o superior).
Además, esta tecnología presenta la ventaja de actualizarse fácilmente mediante la introducción de nuevas secuencias de interés (por ejemplo, nuevos potyvirus, etc.). Además, el uso de esta nueva GP, junto con otras técnicas moleculares que permiten la identificación de las especies de potyvirus correspondientes (por ejemplo, métodos de RT-PCR o HTS basados en cebadores degenerados, etc.) podría ser una buena combinación de métodos para detectar tanto cualquier aislado de potyvirus que se haya descrito previamente o incluso para los que están todavía sin caracterizar
Referencias bibliográficas
- Aparicio, F., Soler, S., Aramburu, J., Galipienso, L., Nuez, F., Pallás, V., & López, C. (2009). Simultaneous detection of six RNA plant viruses affecting tomato crops using a single digoxigenin-labelled polyprobe, 123(1), 117–123. http://dx.doi.org/10.1007/s10658-008-9347-5
- Campanella, J. J., Bitincka, L., & Smalley, J. (2003). MatGAT: An application that generates similarity/identity matrices using protein or DNA sequences. BMC Bioinformatics, 4(1), 29. doi:10.1186/1471-2105-4-29
- Cohen, O., Batuman, O., Stanbekova, G., Sano, T., Mawassi, M., & Bar-Joseph, M. (2006). Construction of a multiprobe for the simultaneous detection of viroids infecting citrus trees. Virus Genes, 33(3), 287–292. doi:10.1007/s11262-006-0067-7
- Herranz, M. C., Sánchez-Navarro, J. A., Aparicio, F., & Pallás, V. (2005). Simultaneous detection of six stone fruit viruses by non-isotopic molecular hybridization using a unique riboprobe or “polyprobe.” Journal of Virological Methods, 124(1–2). doi:10.1016/j.jviromet.2004.11.003
- Jeong, J., Ju, H. & Noh, J. (2014). A Review of Detection Methods for the Plant Viruses. Research in Plant Disease, 20(3), 173–181. doi:10.5423/rpd.2014.20.3.173
- Jordan, R., & Hammond, J. (1991). Comparison and differentiation of potyvirus isolates and identification of strain-specific, virus-specific, subgroup-specific and potyvirus group-common epitopes using monoclonal-antibodies. Journal of General Virology, 72, 25–36. doi:10.1099/0022-1317-72-1-25
- Kumar, S., Stecher, G., Li, M., Knyaz, C., & Tamura, K. (2018). MEGA X: Molecular evolutionary genetics analysis across computing platforms. Molecular Biology and Evolution, 35(6), 1547–1549. doi:10.1093/molbev/msy096
- Lin, L., Li, R., Mock, R., & Kinard, G. (2011). Development of a polyprobe to detect six viroids of pome and stone fruit trees. Journal of Virological Methods, 171(1), 91–97. doi:10.1016/j.jviromet.2010.10.006
- Lopez, M. M., Bertolini, E., Olmos, A., Caruso, P., Gorris, M. T., Llop, P., et al. (2003). Innovative tools for detection of plant pathogenic viruses and bacteria. International Microbiology, 6(4), 233–243. doi:10.1007/s10123-003-0143-y
- MacKenzie, D. J., McLean, M. A., Mukerji, S., & Green, M. (1997). Improved RNA extraction from woody plants for the detection of viral pathogens by reverse transcription-polymerase chain reaction. Plant Disease, 81(2), 222–226. isi:A1997XN27800020
- Minutillo, S. A., Mascia, T., & Gallitelli, D. (2012). A DNA probe mix for the multiplex detection of ten artichoke viruses. European Journal of Plant Pathology, 134(3), 459–465. doi:10.1007/s10658-012-0032-3
- Pallas, V., Mas, P., Sánchez-Navarro, J. A., Pallás, V., Más, P., & Sánchez-Navarro, J. A. (1998). Detection of plant RNA viruses by nonisotopic dot-blot hybridization. Plant Virology Protocols, 81, 461–468.
- Peiro, A., Pallas, V., Angel Sánchez-Navarro, J., Peiró, A., Pallás, V., Sánchez-Navarro, J. A., et al. (2012). Simultaneous detection of eight viruses and two viroids affecting stone fruit trees by using a unique polyprobe. European Journal of Plant Pathology, 132(4), 469–475. doi:10.1007/s10658-011-9893-0
- Saade, M., Aparicio, F., Sánchez-Navarro, J. A., Herranz, M. C., Myrta, A., Di Terlizzi, B., & Pallas, V. (2000). Simultaneous detection of the three ilarviruses affecting stone fruit trees by nonisotopic molecular hybridization and multiplex reverse-transcription polymerase chain reaction. Phytopathology, 90(12). doi:10.1094/PHYTO.2000.90.12.1330
- Saldarelli, P., Barbarossa, L., Grieco, F., & Gallitelli, D. (1996). Digoxigenin-labeled riboprobes applied to phytosanitary certification of tomato in Italy. Plant Disease, 80(12), 1343–1346. isi:A1996VV46100007
- Sánchez-Navarro, J. A. A., Aparicio, F., Rowhani, A., & Pallás, V. (1998). Comparative analysis of ELISA, nonradioactive molecular hybridization and PCR for the detection of prunus necrotic ringspot virus in herbaceous and Prunus hosts. Plant Pathology, 47(6), 780–786. doi:10.1046/j.1365-3059.1998.00301.x
- Sánchez-Navarro, J. A., Canizares, M. C., Cano, E. A. A., Pallas, V., Sánchez-Navarro, J. A., Cañizares, M. C., et al. (1999). Simultaneous detection of five carnation viruses by non-isotopic molecular hybridization. Journal of Virological Methods, 82(2), 167–175. doi:10.1016/s0166-0934(99)00097-x
- Sánchez-Navarro, J. A., Cañizares, M. C., Cano, E. A., & Pallás, V. (1999). Simultaneous detection of five carnation viruses by non-isotopic molecular hybridization. Journal of Virological Methods, 82(2), 167–175. doi:10.1016/S0166-0934(99)00097-X
- Scholthof, K.-B. G., Adkins, S., Czosnek, H., Palukaitis, P., Jacquot, E., Hohn, T., et al. (2011). Top 10 plant viruses in molecular plant pathology. Molecular Plant Pathology, 12(9), 938–954. doi:10.1111/j.1364-3703.2011.00752.x
- Torchetti, E. M., Navarro, B., & Di Serio, F. (2012). A single polyprobe for detecting simultaneously eight pospiviroids infecting ornamentals and vegetables. Journal of Virological Methods, 186(1–2), 141–146. doi:10.1016/j.jviromet.2012.08.005
- Zamora-Macorra, E. J., Ochoa-Martínez, D. L., Valdovinos-Ponce, G., Rojas-Martínez, R., Ramírez-Rojas, S., Sánchez-Navarro, J. Á., et al. (2015). Simultaneous detection of Clavibacter michiganensis subsp michiganensis, Pepino mosaic virus and Mexican papita viroid by non-radioactive molecular hybridization using a unique polyprobe. European Journal of Plant Pathology, 143(4), 779–787. doi:10.1007/s10658-015-0729-1
- Zhang, Z., Peng, S., Jiang, D., Pan, S., Wang, H., & Li, S. (2012). Development of a polyprobe for the simultaneous detection of four grapevine viroids in grapevine plants. European Journal of Plant Pathology, 132(1), 9–16. doi:10.1007/s10658-011-9856-5