Uso de herramientas biotecnológicas en la mejora contra las enfermedades de judía: antracnosis y grasa
Judía común
La judía común (Phaseolus vulgaris L.) es una de las leguminosas más importantes en el consumo humano en semilla o vaina (FAOSTAT, http://faostat.fao.org/). Es una especie extraordinariamente polimórfica, lo que ha permitido que exista un amplio catálogo de variedades tradicionales y comerciales fruto de trabajos de mejora genética. En España, las producciones de algunas variedades tradicionales están amparadas por marcas de calidad diferenciada como Indicaciones Geográficas Protegidas y se puede encontrar un amplio catálogo de variedades en la lista de variedades comerciales de la Oficina Española de Variedades Vegetales (http://magrama.gob.es/).
La susceptibilidad a enfermedades es uno de los principales factores limitantes del cultivo. Al menos 73 enfermedades diferentes pueden afectar a la judía (Schwartz et al. 2005) siendo la antracnosis y la grasa de las más frecuentes, tanto a nivel mundial como en los cultivos españoles. Para su control se proponen diferentes medidas culturales, como la utilización de semillas libres de patógeno, desinfección del material de siembra, rotación de cultivos, eliminación de restos de cosecha, aplicación de productos fitosanitarios, etc (Schwartz et al. 2004).

Sin embargo, el número de materias activas autorizadas para el control de enfermedades en los cultivos cada vez es más limitado en la UE y, además, hay una creciente demanda de los consumidores por unos productos medioambientalmente saludables que rechazan el uso de estas materias activas. Por ello, el uso de resistencias genéticas, siempre que sea posible, es quizás la alternativa más eficaz en el medio plazo. En el caso de antracnosis y grasa, la respuesta a la interacción huésped-patógeno es raza-específica de modo que cada gen de resistencia protege frente a un determinado juego de razas o variantes del patógeno. Se han descrito algunos genes de resistencia, mayoritariamente dominantes (genes Co- y Pse-), frente a los patógenos que causan estas dos enfermedades y alguno de ellos ha sido utilizado con éxito en el desarrollo de nuevas variedades. La mejora genética tradicional para resistencia a estas enfermedades se ha basado en la selección fenotípica de plantas resistentes identificadas mediante tests de resistencia, bajo condiciones controladas o en cultivos en campo naturalmente infectados. En cuanto a los métodos de mejora más usuales para el desarrollo de variedades resistentes se encuentran el método de retrocruzamientos, para incorporar uno o varios genes en una variedad élite, y los métodos selección genealógica o ‘single seed descent’, para agrupar en un genotipo diferentes genes de resistencia. Estos métodos han sido utilizados con éxito en judía, por ejemplo, para incorporar resistencia a antracnosis y a virosis en variedades de la clase comercial 'faba granja asturiana' (Ferreira et al. 2012).
Herramientas biotecnológicas para la mejora
Selección asistida por marcadores
Un marcador molecular se considera cualquier variación, polimorfismo o diferencia a nivel molecular entre dos o más individuos, siendo la molécula más comúnmente analizada el ADN (ácido desoxirribonucleico). La variación observable en un marcador molecular de ADN puede ser debida a diferencias en el número de pares de bases que constituyen ese fragmento y/o a la secuencia de nucleótidos. Los marcadores moleculares, generalmente, se localizan en un único punto del genoma, señalándolo, etiquetándolo o marcándolo, y se heredan como un gen. Tradicionalmente, la posición de un marcador en un cromosoma y su relación con otros marcadores se estima a través de los mapas genéticos de ligamiento, que son una representación de la disposición relativa de diferentes loci en los cromosomas basada en las frecuencias de recombinación entre esos loci. En judía se han publicado varios mapas genéticos de ligamiento que contienen diferentes tipos de marcadores ligados a genes de resistencia frente a diferentes enfermedades, entre ellas antracnosis y bacteriosis del halo.
La selección asistida por marcadores usa el polimorfismo en marcadores moleculares para inferir el fenotipo de un genotipo dado, esto es, para identificar o seleccionar indirectamente plantas portadoras de determinados caracteres a partir de la información proporcionada por los marcadores (Collard y Mackill 2008). En judía común se han publicado marcadores ligados a diferentes genes o QTLs (quantitative trait loci) implicados en la expresión de caracteres importantes, como los genes de resistencia antracnosis y a grasa (véase lista de marcadores en la web de la Bean Improvement Cooperative, http://bic.css.msu.edu/). Sin embargo, la existencia de marcadores ligados a un gen de interés no necesariamente implica que ese marcador pueda ser utilizado en un programa de mejora particular. Su utilidad dependerá de la detección de polimorfismo entre genotipos, el nivel de ligamiento (distancia genética), el tipo de ligamiento (acoplamiento & repulsión) el tipo de herencia (dominante & codominante) o el tipo de marcador. De ahí la importancia de disponer de una batería de marcadores ligados a un gen de interés.
Genoma de referencia
El genoma es la secuencia de nucleótidos en los diferentes cromosomas de una especie incluyendo los genes, su estructura y función. El genoma de judía común ha sido publicado recientemente con un total de 27197 genes automáticamente anotados y distribuidos en 11 cromosomas (Schmutz et al. 2014). La disponibilidad de este genoma de referencia (www.phytozome.net) ha permitido:
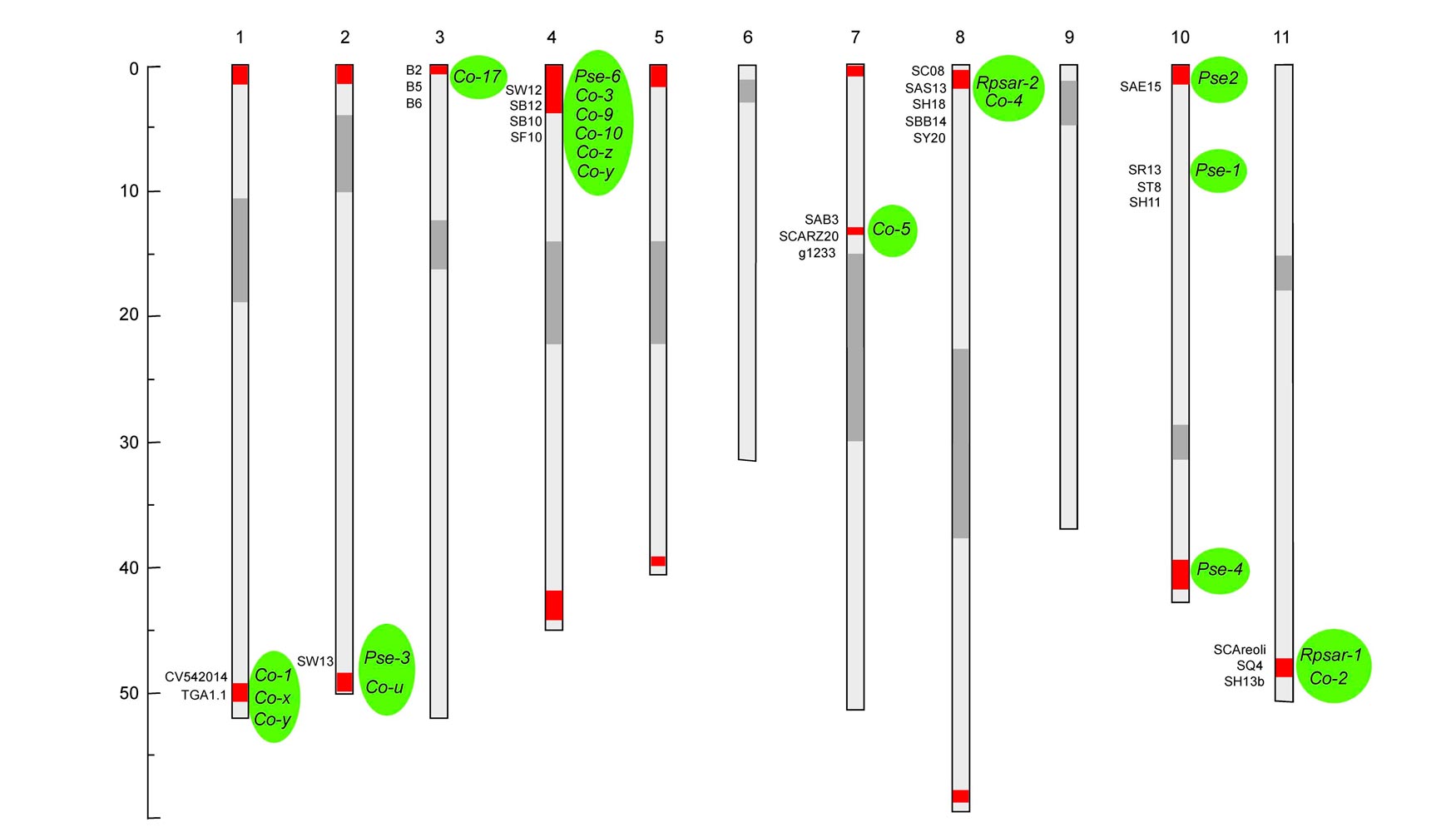
- Conocer la posición física (situación en un cromosoma) de los marcadores moleculares mediante un alineamiento (BLAST, basic local alignment search tool) de la secuencia del marcador con la secuencia del genoma de la especie. De este modo, es posible conocer la región física donde se localizan genes mayores a partir de las coordenadas de los marcadores moleculares descritos como ligados. Por ejemplo, los marcadores tipo SCAR (sequence characterized amplified regions) SCAreoli y SQ4 estrechamente ligados a genes de resistencia a antracnosis en el cluster Co-2 están localizados, respectivamente, en las coordenadas 47.11 y 48.06 millones de pares de bases del cromosoma 11 (Figura 1).
- Diseñar otros marcadores moleculares 'a la carta' para cada laboratorio y material concreto, basados en el conocimiento de la secuencia de nucleótidos del genoma de referencia. De este modo, también se puede saturar de marcadores una determinada región cromosómica para aproximarse a un gen concreto, o diseñar nuevos marcadores que sean más informativos (p.ej. pasar de un marcador dominante a uno codominante) que los disponibles. Para ello, existen una serie de programas informáticos que ofrecen la posibilidad de desarrollar otros marcadores como microsatélites, Indels (insertion-deletion) o SCARs (p.ej. BatchPrimer3, OligoAnalyzer 3.1, etc.).
- Identificar el gen, entendido como la secuencia de nucleótidos, responsable de la expresión de un carácter como puede ser la resistencia a un patógeno, lo que se conoce como análisis genético directo (forward genetic). Una vez identificado el gen, se puede desarrollar un marcador interno, preferentemente a partir de las regiones de ese gen que se traducen (exones) para disponer de un marcador funcional del propio gen. La selección asistida con marcadores funcionales supone ausencia de recombinación entre el gen y el marcador (excepto posible recombinación intragénica) y por tanto aumenta la eficiencia del programa de mejora.
- Conocer la organización de los genes en el cromosoma. Así, del total de genes predichos en judía, 376 codifican para dominios asociados con la respuesta a patógenos (genes R). Se ha observado que la mayor parte de los genes R se encuentran agrupados formando lo que se conoce como clusters de genes, localizados en muchos casos en los telómeros de los cromosomas (Meziadi et al. 2016). Por ejemplo, en la región de los 47-48 Mpb del cromosoma 11, donde se han localizado el cluster Co-2 de resistencia a antracnosis y el gen Rpsar-1 de resistencia a grasa, se han identificado unos 60 genes R, y cualquiera de ellos es candidato a estar controlando la resistencia a un determinado patógeno o variante patogénica (Figura 1).
Genotipado masivo
Si se entiende por genotipo el conjunto de genes de un organismo, el genotipado es el conocimiento detallado de tales genes. En contraposición, el fenotipo es el conjunto de rasgos, caracteres o atributos de un organismo y el fenotipado es el conocimiento detallado los caracteres de un organismo. Teniendo en cuenta que los marcadores moleculares etiquetan genes o puntos del genoma, el concepto genotipado masivo (a gran escala) actualmente se suele aplicar para definir la variación para marcadores que hay entre individuos, siendo los polimorfismos de una sola base (SNPs, single nucleotide polymorphisms) los más usados. En los últimos años, se han descrito diferentes tecnologías, agrupadas bajo el término NGS (next generation sequencing), que permiten conocer la secuencia de fragmentos de ADN de una manera rápida y económica frente a las técnicas tradicionales basadas en el método Sanger. Basándose en las NGS se han publicado métodos y estrategias que permiten un genotipado masivo, como el método de genotyping by sequencing (GbS, Elshire et al. 2011) en el que, simplificadamente, el ADN es cortado con un enzima de restricción, los fragmentos obtenidos son secuenciados y las secuencias son alineadas para detectar variantes en la posición de un nucleótido. Por ejemplo, utilizando GbS hemos obtenido 50.504 marcadores SNPs con una posición física conocida en un juego de 24 genotipos de judía común. A pesar del elevado número de marcadores proporcionados con estas tecnologías, también existen limitaciones, como que los marcadores SNPs no se distribuyen homogéneamente en los cromosomas, habiendo regiones muy saturadas de marcadores, generalmente los extremos de los cromosomas o telómeros, y otras con pocos marcadores, que coinciden con las zonas centroméricas del cromosoma.
Otra tecnología que permite el genotipado a gran escala son los chip de ADN para genotipado (DNA microarray) que son matrices que permiten conocer las secuencias de un genoma en determinadas posiciones. Por ejemplo, en judía se ha desarrollado el chip BARCBean6K_3 BeadChip que incluye 5398 SNPs con posición física conocida (Song et al. 2015). Este chip nos ha permitido obtener 2561 SNPs en la población de líneas recombinantes de judía XanaxCornell49242 de los que 1912 no fueron considerados debido a que presentaban una segregación distorsionada o a que su posición en el genoma era coincidente con la de otros SNPs (redundancias). La distribución en la mapa genético de estos SNPs no fue homogénea, agrupándose en las regiones teloméricas y subteloméricas.
Tanto los chip de ADN como las técnicas NGS puede ser aplicadas al estudio del transcriptoma, esto es, las secuencias transcritas de los genes y su cantidad (nivel de expresión) en un estado fisiológico o tejido determinado de la planta. Así, por ejemplo, el análisis del transcriptoma mediante su secuenciación (RNA-Seq; Wang et al. 2009) comienza a ser utilizado en la identificación de la batería de genes que participan en la respuesta a la interacción huésped-patógeno para, combinando los datos genéticos y genómicos, llegar a identificar los genes mayores responsables de la resistencia. Los estudios del transcriptoma están, además, desvelando nuevos mecanismos de regulación de la información contenida en el ADN, como la metilación o el splicing alternativo (varios transcritos de un mismo gen).
Aplicación del genotipado masivo
El genotipado a gran escala ofrece la posibilidad de desarrollar mapas genéticos de alta densidad en poblaciones segregantes que permiten el estudio de la herencia de caracteres y la localización precisa de genes o QTLs (análisis genético directo). Este genotipado está siendo utilizado en estudios de asociación en paneles para la identificación de nuevos genes o regiones implicadas en el control de caracteres (GWAS, genomic wide association studies). También ha sido aplicado en estudios de diversidad que permiten conocer la estructura o grupos de germoplasma, y en la diferenciación de variedades, particularmente en aquellos casos con fenotipos muy similares. Por ejemplo, el polimorfismo revelado por un análisis GbS ha permitido disponer de numerosos SNPs que diferencian el juego de 16 lineas isogénicas del tipo faba granja (todas con idéntico fenotipo de semilla) que disponen de diferentes combinaciones de genes introgresados (Ferreira et al. 2012). La mayoría de estos marcadores diferenciadores se localizan en las regiones que contienen los genes introgresados. Finalmente, este genotipado masivo puede ser usado para la selección genómica, una forma de selección asistida que considera simultáneamente muchos marcadores para estimar el valor fenotípico de un genotipo y, de este modo, acelerar los programas de mejora (Heffner et al. 2009). Obviamente, la eficiencia de la selección genómica guarda estrecha relación con el conocimiento del efecto de cada marcador en el fenotipo por lo que se recomiendan estudios previos en poblaciones prueba. Este tipo de selección puede ser especialmente útil en programas de mejora en los que se manejan caracteres con un control genético complejo, como los controlados por varios QTL, cuyo efecto es difícil de valorar. También la selección genómica ha sido propuesta para acelerar la introgresión de varios genes mayores en un cultivar élite a través de un programa de retrocruzamientos. En todo caso, es recomendable evaluar el coste y ganancia por unidad de tiempo que proporciona la selección genómica frente a la selección fenotípica o la selección asistida por pocos marcadores.
Antracnosis
La antracnosis en judía común está causada por el hongo Colletotrichum lindemuthianum. (Sacc. y Magn.) Lams.-Scrib. Es una enfermedad extendida a nivel mundial y se considera grave, ya que puede llegar a causar pérdidas del 100% de la producción (Singh y Schwartz 2010). Esta enfermedad está particularmente presente en regiones con temperaturas moderadas y elevada humedad relativa. En condiciones favorables, las conidias germinan y penetran en los tejidos para, con el tiempo, producir necrosis en los tejidos infectados. Los síntomas de la enfermedad pueden aparecer en todos los órganos aéreos de la planta. Comienzan con pequeñas lesiones sobre el tallo y sobre las venas de las hojas, siendo más frecuentes en el envés que en el haz, en forma de chancros de color pardo o rojizo (Figura 2A).
Cuando la enfermedad progresa, en las lesiones se producen conidias que son fácilmente dispersadas en gotas de lluvia para infectar otras plantas. Estas conidias pueden permanecer en el suelo por varios años. Si la infección afecta a estadíos jóvenes de la planta puede causar su muerte (Figura 2B). En planta adulta la infección llega a las vainas (Figura 2C) y de aquí pasa a las semillas, produciendo unas manchas oscuras especialmente visibles en semillas de color blanco (Figura 2D), lo que devalúa totalmente el precio de la cosecha. La semilla infectada es un mecanismo de transmisión de la enfermedad junto con los restos de cosecha.




Este hongo es extremadamente variable, describiéndose más de 100 razas o variantes patogénicas diferentes en base a la respuesta de 12 variedades diferenciales de judía. Hasta la fecha, se ha constatado la presencia de las razas 3, 6, 19, 38 y 102 de antracnosis en el norte de España (Ferreira et al. 2008). En judía se han descrito más de 20 genes de resistencia diferentes, denominados genes Co- (Co-1 a Co-17; Co-u, Co-v, Co-w, Co-y y Co-z), la gran mayoría de ellos dominantes. Tradicionalmente, se consideraba la existencia de variantes alélicas en algunos de estos genes (loci Co-1, Co-3, Co-4) para explicar las diferencias en el espectro de resistencia de variedades que, presumiblemente, compartían un mismo gen (Ferreira et al. 2013). Sin embargo, diferentes análisis genéticos y la secuenciación del genoma de judía han puesto de manifiesto una organización de los genes de resistencia a antracnosis en grupos de genes estrechamente ligados entre ellos, formando lo que se denomina cluster de genes, donde diferentes genes confieren resistencia a diferentes razas. Hasta el momento, se han identificado siete principales regiones cromosómicas o clusters que controlan la resistencia genética a antracnosis en judía, cada una de ellas etiquetada con numerosos marcadores moleculares ligados (Figura 1).
Bacteriosis del halo o grasa
La grasa de las judías está causada por la bacteria Pseudomonas syringae pv. phaseolicola (Psp). Este patógeno presenta una distribución mundial y constituye un factor limitante del rendimiento de este cultivo en muchas zonas productoras. En España, puede decirse que está generalizada en toda la Península, pero es más grave en zonas de clima húmedo con temperaturas entre moderadas y frías. La severidad de esta enfermedad varía de unos años a otros dependiendo de las condiciones ambientales, estimándose pérdidas de rendimiento entre el 20 y el 60% (Schwartz 1989). En cuanto a la variación patogénica de esta bacteria, Taylor et al. (1996) describieron 9 razas fisiológicas basándose en la respuesta de 8 variedades diferenciales de judía habiéndose constatado la presencia de las razas 6 y 7 en los cultivos locales. Sin embargo, posteriormente se han identificado aislados de Psp que no se corresponden con ninguno de los patrones de raza descritos y presentan patrones de reacción variados (Rico et al. 2003), sugiriendo la existencia de una mayor variabilidad del patógeno.
Esta bacteria aprovecha heridas, roturas o estomas para penetrar en los tejidos y desarrollarse. Los primeros síntomas foliares aparecen como manchas acuosas que se agrandan y frecuentemente se unen con lesiones adyacentes (Figura 3A). Después, Psp produce un halo de tejido clorótico alrededor de las manchas acuosas. Estas manchas de aspecto grasiento terminan por necrosarse, produciendo una disminución del rendimiento e incluso la muerte de la planta. Las lesiones de las vainas aparecen al principio como manchas acuosas más o menos redondeadas que se pueden agrandar y tornar oscuras, rojas y ligeramente hundidas (Figura 3B). La infección puede tener lugar en los elementos vasculares de la sutura ventral y dorsal, produciendo remojado en los tejidos adyacentes. La infección a menudo sigue los elementos vasculares de las suturas y penetra en las semillas a través del fonículo. Los síntomas en las semillas se manifiestan como manchas de color amarillo mantequilla, más evidentes en las de color blanco. Las plántulas que se desarrollan a partir de semillas severamente infectadas pueden tener daños en su ápice de crecimiento, presentar enanismo y morir (Figura 3C).



Se han descrito varios genes de resistencia con una herencia mendeliana, similar a la descrita en antracnosis (genes Pse-1 a Pse-6, Rpsar-1 y Rpsar-2). Algunos de estos genes de resistencia se han localizado directamente en el mapa genético de la especie, o indirectamente a través de marcadores moleculares ligados (Singh y Schwartz 2010; Miklas et al. 2014; Chen et al. 2010), y muchos de ellos se localizan en las mismas regiones donde se ha identificado genes de resistencia a antracnosis (Figura 1). Sin embargo, la resistencia a este patógeno parece ser más compleja de lo que ha sido descrito hasta el momento en los trabajos clásicos, ya que, en ausencia de alguno de estos genes mayores, se ha identificado una resistencia de tipo cuantitativo y se han localizado diferentes QTLs implicados en la respuesta (Trabanco et al. 2014).
Perspectivas de futuro
Aunque resulta muy difícil predecir el futuro, es razonable prever en el medio plazo:
- Una mayor reducción del coste de tecnologías genómicas de alto rendimiento (p.ej. chips, NGS) de modo que sea posible secuenciar y anotar muchos genotipos en un coste razonable.
- Un mayor desarrollo de herramientas bioinformáticas, como programas informáticos y paquetes estadísticos, que permitan manejar y aprovechar toda la información generada a un amplio abanico de usuarios.
- Un rápido incremento en el conocimiento del papel de los genes automáticamente anotados (por anotación manual y resecuenciaciones), así como en las redes de genes que participan en la expresión de los fenotipos como la resistencia a enfermedades, lo que permitirá disponer de una nueva generación de marcadores funcionales que ayuden en la selección genómica.
- Una automatización del fenotipado que minimice la componente subjetiva en la valoración de muchos caracteres, de manera que se pueda establecer una mejor asociación de los marcadores a los caracteres y a los genes que los controlan.
- Un desarrollo de las bases de datos que incluyan datos fenotípicos, genéticos, genómicos, transcriptómicos y metabólicos de las que se pueda extraer información útil para los mejoradores.
- El uso de la cisgénesis o transgénesis como herramienta en el desarrollo de nuevos cultivares de judía o análisis genéticos y genómicos.
Agradecimientos
Los autores agradecen al INIA, Ministerio de Economía y Competitividad, Madrid, la financiación de los trabajos de mejora genética de judía a través de diferentes proyectos de investigación (RTA2009-0093, RTA2011-0076-CO2-01, RTA2012-0056), así como la financiación de un contrato DOC-INIA-CCAA (A. Campa) y una beca FPI-INIA (E. Murube).
Referencias
- Chen N.W.G., Sevignac M., Thareau V., Magdelenat G., David P., Ashfield T., et al. 2010. Specific resistances against Pseudomonas syringae effectors AvrB and AvrRpm1 have evolved differently in common bean (Phaseolus vulgaris), soybean (Glycine max), and Arabidopsis thaliana. New Phytol. 187:941-956
- Collard B.C.Y., Mackill D.J. 2008. Marker-assisted selection: an approach for precision plant breeding in the twenty-first century. Phil. Trans. R. Soc. B. 363:557–572
- Elshire R.J., Glaubitz J.C., Sun Q., Poland J.A., Kawamoto K., Buckler E.S., Mitchell E. 2011. A robust, simple genotyping-by-sequencing (GBS) approach for high diversity species. PLoS ONE 6: e19379. doi:10.1371/journal.pone.0019379
- Ferreira J.J, Campa A., Kelly J.D. 2013. Organization of genes conferring resistance to anthracnose in common bean. In: Genomics Applications in Plant Breeding, Editors: R. Tuberosa and R. Varshney, Wiley-Blackwell Pubs 151-182
- Ferreira J.J., Campa A., Pérez-Vega E. 2008. Reaction of a bean germplasm collection against five races of Colletotrichum lindemuthianum identified in northern Spain and implications for breeding. Plant Dis. 92:705-708
- Ferreira J.J., Campa A., Pérez-Vega E., Rodríguez-Suárez C., Giraldez R. 2012. Introgression and pyramiding into common bean market class fabada of genes conferring resistance to anthracnose and potyvirus. Theor. App. Genet. 124:777-88
- Heffner E.L., Sorrells M.E., Jannink J.L. 2009. Genomic selection for crop improvement. Crop Sci. 49:1-12
- Meziadi C., Richard M.M.S., Derquennes A., Thareau V., Blanchet S., Gratias A., Pflieger S., Geffroy V. 2016. Development of molecular markers linked to disease resistance genes in common bean based on whole genome sequence. Plant Sci. 242:351-357
- Miklas P.N., Fourie D., Trapp J., Davis J., Myers J.R. 2014. New loci including pse-6 conferring resistance to halo bacterial blight on chromosome Pv04 in common bean. Crop Sci. 54:2099–2108
- Rico A., López R., Asensio C., Aizpún M., Asensio-Manzanera M.C., Murillo J., et al. 2003. Nontoxigenic strains of P. syringae pv. phaseolicola are the main cause of halo blight of beans in Spain and scape current detection methods. Phytopathology 93:1553-1559
- Schmutz J., McClean P.E., Mamidi S., Wu G.A., Cannon S.B., et al. 2014. A reference genome for common bean and genome-wide analysis of dual domestications. Nat. Genet. 46:707-713
- Schwartz H.F. 1989. Añublo de Halo. En: Problemas de Producción del Fríjol en los Trópicos (eds. M. Pastor-Corrales, H.F. Schwartz). CIAT, Cali (Colombia), pp. 331-349. ISBN: 958-9183-78-6
- Schwartz H.F., Brick M.A., Harveson R.M., Franc G.D. 2004. Dry Bean Production & Integrated Pest Management. 2nd edition. Regional publication by Central High Plains Dry Bean and Beet Group, Colorado State University, University of Nebraska, and University of Wyoming: Bulletin 562A. 167 p
- Schwartz H.F., Pastor-Corrales M.A. 2005. Anthracnose. In: Compendium of Bean Diseases. Edited by Schwartz H.F., Steadman J.R., Hall R., Forster R.; APS Press; pp 25-27
- Singh S.P., Schwartz H.F. 2010. Breeding common bean for resistance to diseases: a review. Crop Sci. 50:2199-2223
- Song Q., Jia G., Hyten D.L., Jenkins J., Hwang E.-Y., et al. 2015. SNP assay development for linkage map construction, anchoring 8 whole genome sequence and other genetic and genomic applications in common bean. G3, Genes, Genomes, Genetics 5:2285-2290
- Taylor J.D., Teverson D.M., Allen D.J., Pastor-Corrales M.A. 1996. Identification and origin of races of Pseudomonas syringae pv. phaseolicola from Africa and other bean growing areas. Plant Pathol. 45:469–478
- Trabanco N., Asensio-Manzanera MC, Pérez-Vega E., Ibeas A., Campa A., Ferreira J.J. 2014. Identification of QTLs involved in the response of common bean to Pseudomonas syringae pv phaseolicola. Mol. Breed. 33:577-588
- Wang Z., Gerstein M., Snyder M. 2009. RNA-Seq: a revolutionary tool for transcriptomics. Nature Rev. 10:57-63