Mejora genética de resistencia a enfermedades en judía común
Las enfermedades son uno de las principales causas de pérdidas en los cultivos. En una agricultura moderna y medioambientalmente responsable, se tiende hacia el uso limitado de productos químicos para el control de las enfermedades. En este sentido, la mejora genética vegetal ha ofrecido y puede ofrecer soluciones mediante el desarrollo de variedades resistentes a enfermedades. En este texto, brevemente, se revisan las principales enfermedades que afectan al cultivo de la judía común, así como las herramientas disponibles para la mejora genética de resistencia a enfermedades en esta especie.
Introducción
Las plantas cultivadas conviven con multitud de microorganismos como virus, bacterias, hongos o nematodos. En ocasiones, la interacción con determinados microorganismos produce daños visibles en las plantas. Una enfermedad no es más que una alteración del normal desarrollo de una planta a consecuencia de la interacción prolongada entre la planta y un microorganismo en unas condiciones ambientales favorables.
La judía común (Phaseolus vulgaris L.) es una leguminosa que ha sido domesticada por los pueblos precolombinos en dos zonas del continente americano, en la región andina y mesoamericana. Es una especie extremadamente diversa en cuanto a fenotipo de planta, vaina o semilla, lo que se ha traducido en la posibilidad de diversas formas de aprovechamiento, métodos de cultivo y rango de nichos a los que puede adaptarse (Voysest 2000). Según la variedad o cultivar, esta especie puede ser utilizada en el consumo humano por sus vainas (judía de verdeo o ‘snap beans’) o por sus semilla (judía grano o ‘dry beans’), que pueden ser consumidas tras un proceso de tostado o cocerse directamente después de un proceso de rehidratación. A nivel mundial, la mayor superficie se destina a la producción de semilla seca. En el año 2012 se produjeron 23,9 y 20,7 millones de toneladas de semilla seca y vaina, respectivamente, destinándose a su cultivo 30,8 millones de ha (FAOSTA, http://faostat.fao.org/).
Según esta misma fuente, en 2012 en España la mayor parte de la superficie que se destinó a este cultivo fue para uso en verdeo; 9.900 ha de un total de 16.500 ha. No obstante, también hay importantes producciones de judía grano entre las que se incluyen variedades tradicionales de gran prestigio como los tipos faba granja, verdina, riñón, canellini, canela, ganxet, gernikesa, etc. Algunas de estas producciones están amparadas por marcas de calidad diferenciada como Indicaciones Geográficas Protegidas (véase, por ejemplo http://www.magrama.gob.es/).
Principales enfermedades en los cultivos de judía
La judía común puede ser atacada por numerosos microorganismos causando una amplia diversidad de enfermedades. En judía han sido descritas al menos 73 enfermedades diferentes causadas por hongos, bacterias, virus, nematodos o fitoplasmas y que atacan las raíz o la parte aérea de la planta (Schwartz et al. 2005). La incidencia de éstas en los cultivos depende del área de cultivo, el manejo del mismo y de los cultivares utilizados. Las principales enfermedades citadas a nivel mundial se resumen en la Tabla 1.
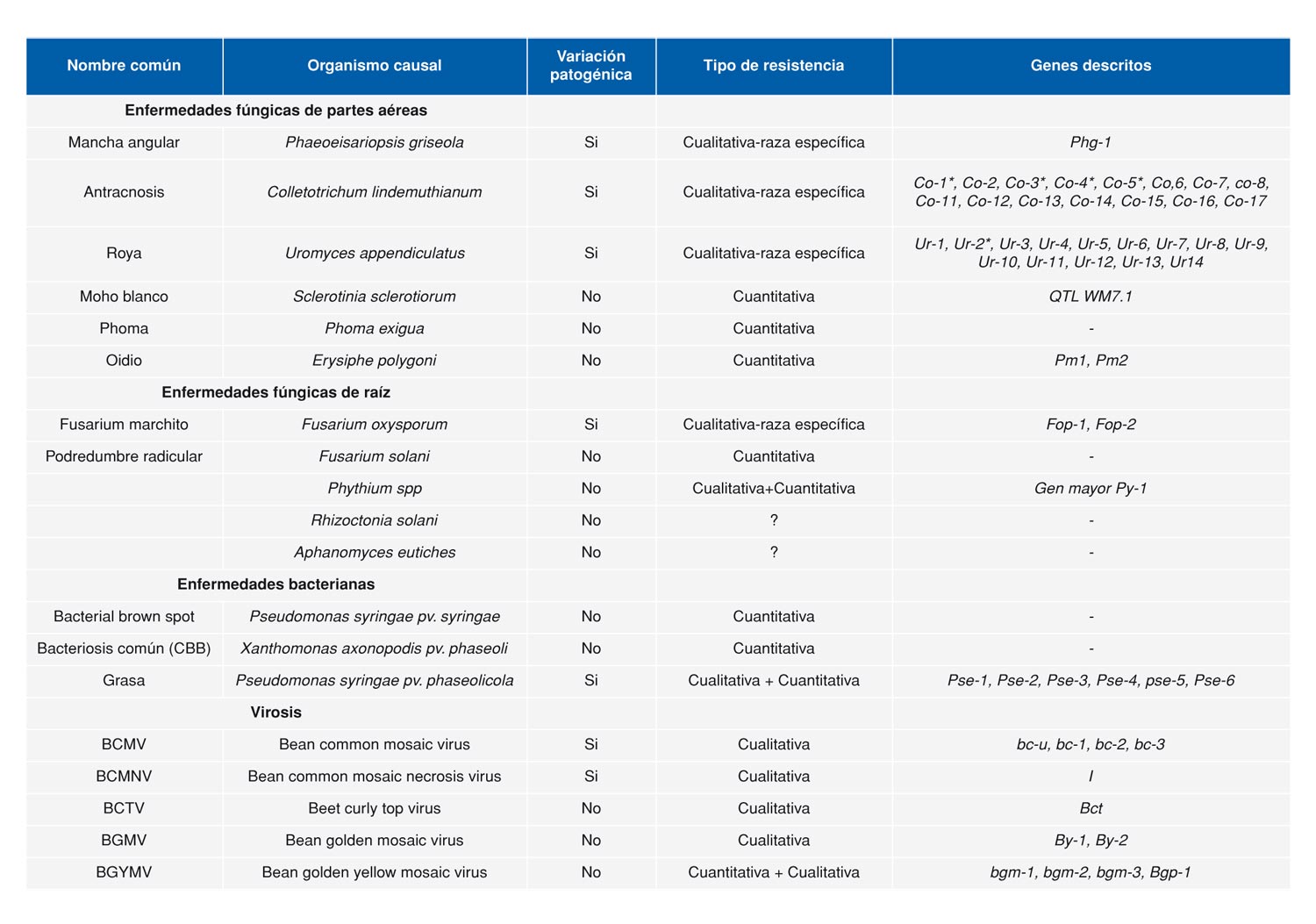
Tabla 1. Lista de las principales enfermedades presentes en los cultivos de judía común (Phaseolus vulgaris L.) (adaptada de Singh y Schwartz 2010).
En España son frecuentes enfermedades como el virus del mosaico común y necrótico de la judía (BCMV y BCMNV), antracnosis, grasa y podredumbres radiculares aunque también se han detectado algunas enfermedades emergentes como el oidio, el moho blanco, o ascochyta (Figura 1). La presencia de alguna de estas enfermedades ya fue citada por Puerta Romero (1962) en los cultivos españoles a mediados del siglo XX.

Tipos de resistencia genética a enfermedades en plantas
Atendiendo al modo de herencia en la respuesta a la interacción huésped-patógeno se pueden diferenciar dos tipos principales de resistencia genética:
- Resistencia de naturaleza cualitativa. La respuesta al patógeno muestra claramente diferenciadas en la especie las clases fenotípicas resistente (ausencia o síntomas mínimos) y susceptible (presencia de síntomas extremos). Este tipo de resistencia está controlada por un limitado número de genes cuyo efecto en la respuesta resistente/susceptible es claramente identificable en análisis genéticos clásicos. La mayor parte de los genes de resistencia descritos en judía presentan una dominancia completa, es decir, el alelo dominante del gen es el que confiere la resistencia (véase Lista de genes de judía en http://bic.css.msu.edu/). Son una excepción, por ejemplo, los genes de resistencia a virus del mosaico común y necrótico (véase Tabla 1), ya que la resistencia está conferida por los alelos recesivos de la combinación de diferentes genes (Drijfhout 1978). Generalmente, las resistencias cualitativas están asociadas a los patógenos clasificados como especialistas, para los que se han descrito razas o variantes patogénicas. Estas razas son identificadas por la respuesta diferencial de un juego de variedades de judía. La antracnosis, la roya o la grasa son ejemplos de resistencia-raza específica en judía y en los que se han propuesto juegos de variedades diferenciales estandarizados para la caracterización de las variantes patogénicas. Por ejemplo, las variedades Canadian Wonder, A52 (portadora del alelo de resistencia Pse-4), Tendergreen (Pse-3), UI3 (Pse-1, Pse-4), 1072 (Pse-2), ZAA55 (Pse-3, Pse-4), ZAA12 (Pse-2, Pse-3, Pse-4, pse-5), y Guatemala 196 (Pse-1?, Pse-3, Pse-4) han sido propuestas para la diferenciación de las razas de grasa (Taylor et al. 1996).
- Resistencia de naturaleza cuantitativa. La respuesta al patógeno no muestra claramente diferenciadas las clases fenotípicas resistente y susceptible, sino que se observan diferentes niveles de respuesta o síntomas de la enfermedad en la especie. Esta resistencia está controlada por QTLs (quantitative trait loci), de modo que cada QTL contribuye a una proporción de la expresión de la respuesta resistente frente al patógeno (porcentaje de la varianza fenotípica explicada). Este tipo de resistencia suele estar asociada a los patógenos poco especialistas. En el caso de la judía hay varios ejemplos de este tipo resistencia (véase Tabla 1) como el moho blanco o la bacteriosis causada por X. campestris. Para estos patógenos, se han descrito algunos genotipos de judía que presentan elevados niveles de resistencia y en los que se han caracterizado los QTLs responsables de esta respuesta (Tabla 1).
Mejora genética de resistencia enfermedades
La mejora genética persigue desarrollar plantas con fenotipos superiores, adaptados a las necesidades de los agricultores y consumidores, que sean distintos a los disponibles, homogéneos y con unas características estables. En la práctica, la mejora genética incluye dos pasos principales; aprovechar la variación existente o, en su defecto, generarla mediante cruzamientos, y seleccionar los genotipos con características superiores hasta fijar los caracteres deseados (Moose y Mumm 2008). Sin embargo, en el caso de la mejora genética para resistencia a enfermedades, además de la planta debe tenerse en cuenta la presencia de un segundo actor, el patógeno, ambos dentro de un escenario particular, el ambiente. Tanto el patógeno como la planta pueden mostrar variabilidad lo que se traduce en variación en la respuesta a la interacción entre ambas especies. Aunque la existencia de variación ofrece la posibilidad de selección, ésta debe de ser conocida y heredable para poder ser usada por el mejorador. Para abordar un programa de mejora genética de resistencia a enfermedades conviene que el mejorador disponga de:
- Conocimientos sobre el patógeno responsable de los daños así como su variación patogénica (variante en agresividad o variantes patogénicas). Generalmente se recomienda usar aislamientos locales, es decir, obtenidos en los escenarios en los que se van a cultivar las variedades desarrolladas y genéticamente homogéneos. Sin embargo, la creciente facilidad de intercambios de materiales así como los efectos del cambio climático hace que las poblaciones de patógenos puedan variar en corto espacio de tiempo.
- Métodos para el manejo, multiplicación y conservación del patógeno. Para alguno de los patógenos de judía hay descritos métodos de manejo estandarizados que permite comparar resultados entre laboratorios (Guías Prácticas de Laboratorio para el Manejo de Patógenos de Fríjol en la http://ciat.cgiar.org/).
- Test de resistencia para evaluar la repuesta en la interacción planta-patógeno. Este tipo de ensayos busca reproducir las condiciones para el desarrollo de la enfermedad, esto es, poner en contacto la planta y el patógeno en unas condiciones favorables. Los tests de resistencia se pueden desarrollar en campo en los que las plantas son naturalmente infectadas o en condiciones controladas en cámaras de cultivo o invernaderos. En las resistencias cuantitativas las condiciones ambientales juegan mayor papel en la respuesta de las plantas, de modo que para minimizar sus efectos se tiende a utilizar diseños más complejos en cuanto a número de repeticiones, distribución de las repeticiones y número de ambientes. La Figura 2 muestra, a modo de ejemplo, los pasos seguidos para realizar una inoculación artificial en invernadero frente a moho blanco en plantas de judía.

- Métodos para valorar la variación en la interacción planta-patógeno. En muchas enfermedades es común usar una escala de daños 1-9 (van Schoonhoven y Pastor Corrales 1987) teniendo en cuenta la proporción de la planta afectada, donde 1 es ausencia de daño y 9 muerte de la planta. Para algunas enfermedades es posible cuantificar los daños mediante análisis de imágenes con programas informáticos especializados (p.ej. Assess 2.0).
- Variación en genotipos para la respuesta al patógeno. En este punto se deben incluir las fuentes de resistencia descritas y contrastadas, si están disponibles. Si no se dispone de fuentes de resistencia se puede recurrir a realizar cribados en colecciones de germoplasma como colecciones nucleares. Por ejemplo, una colección nuclear de judías derivada de la colección del Centro de Recursos Fitogenéticos-CRF (Figura 3) fue usada para identificar fuentes de resistencia frente a oidio, un patógeno emergente en los cultivos de judía en el norte de España (Trabanco et al. 2012).

Métodos de mejora genética habituales en judía común
La mejora genética de esta especie seguramente comenzó en Europa a mediados del siglo XIX en empresas o jardines botánicos. La judía común es una especie esencialmente autógama (Ferreira et al. 2000), es decir, se autofecunda. Por ello, las variedades generalmente están constituidos por una o varias líneas homocigotas. La selección del método de mejora más apropiado dependerá de los objetivos concretos, así como de los recursos disponibles por el mejorador. Con ligeras variantes en su desarrollo como la etapa o estrategia en la selección, los métodos de mejora genética más usados en judía son:
Método de selección genealógica o pedigrí. Este método es recomendado cuando se persigue agrupar varios caracteres como resistencias a varios patógenos en un mismo genotipo. Este método consiste en seleccionar parentales con características superiores y obtener una población de plantas F1 a partir de cruzamientos sencillos o compuestos (más de dos parentales). A partir de este punto se desarrollan varias generaciones de autofecundación, seleccionando en cada generación las plantas portadoras de los caracteres deseables (resistentes) para contribuir a la siguiente generación. Con el avance de las generaciones de autofecundación y selección se incrementa la proporción de homocigotos y se fijan los caracteres deseables. Este método ha sido usado para agrupar los genes Co-2 y Co-9 que confieren resistencia frente a las razas locales de antracnosis 6, y 38 en el genotipo de faba granja A2438 (Ferreira et al. 2012).
Método de ‘single seed descent’. Este método es recomendado cuando se persigue recombinar las características, como resistencias a varios patógenos, presentes en dos o más genotipos sin selecciones en fase intermedias. Este método consiste en seleccionar parentales y obtener poblaciones de plantas F1 o F2 a partir de cruzamientos sencillos o compuestos (más de dos parentales). A partir de las plantas F2 se llevan a cabo 4-6 generaciones de autofecundación de modo que en cada generación se autofecunda una planta descendiente de cada plantas F2. Con la finalización de este proceso se dispone de una colección de líneas recombinantes para ser seleccionadas en función de la expresión de los caracteres deseados. Este método ha sido usado para desarrollar poblaciones permanentes de mapeo a la vez que se agrupan genes/QTLs en una misma línea (Figura 4).

Método de retrocruzamientos. Este método es recomendado para la introgresión de un carácter controlado por un limitado número de genes o QTLs en un genotipo o cultivar élite. Este método consiste en seleccionar un donante portador del carácter y cruzarlo con el genotipo que se pretende mejorar (parental recurrente). Las plantas F1 son cruzadas con el parental recurrente para obtener F1Bc1 (primera generación de retrocruzamientos). Seguidamente, las plantas F1Bc1 seleccionadas como portadoras del carácter son cruzadas, con el parental recurrente (retrocruzadas). De este modo se desarrollan varios ciclos de retrocruzamientos hasta alcanzar la 6ª generación de retrocruces donde se espera disponer de plantas prácticamente idénticas al parental recurrente pero portadores del gen/QTL introgresado. Llegado a este punto se desarrollan varias generaciones de autofecundación y selección genealógica para fijar los caracteres deseados controlados por los genes introgresados. Este método ha sido usado para introgresar resistencia a antracnosis (genes Co-2 y Co-9) y a virus del mosaico común (genes I y bc-3) en la líneas faba granja asturiana (Ferreira et al. 2012).
Herramientas biotecnológicas disponibles para la mejora genética en judía
En los últimos 20 años la mejora genética vegetal en judía ha experimentado cambios significativos, principalmente, al disponer de nuevas herramientas y mayor información sobre el control genético de las resistencias en las principales enfermedades. Esto permite al mejorador desarrollar una mejora orientada, conociendo los genes o QTL que maneja. Estos cambios han sido consecuencia, por una parte, del desarrollo de la metodología de amplificación del ADN (PCR, reacción en cadena de la polimerasa). Con ella ha sido posible identificar y etiquetar muchos genes o QTLs de resistencia y localizarlos en el mapa genético de la especie. Toda esta información ha permitido constatar que los genes o QTLs de resistencia están localizados en regiones específicas y que algunas de estas regiones participan en la resistencia frente a diferentes patógenos. Tal es el caso de un extremo del grupo de ligamiento Pv11 donde se mapearon genes de resistencia a antracnosis (gen Co-2), roya (genes Ur-11 y Ur-3) y oidio (gen Pm1). Así mismo, el mejorador ha podido disponer de un abanico de marcadores moleculares ligados a los genes o QTLs que controlan estas resistencias, de modo que tiene la posibilidad de realizar la selección indirecta, esto es, inferir el fenotipo de un individuo a partir de la expresión de un marcador molecular (Figura 5). En judía se han descrito numerosos marcadores ligados a genes de resistencia de muchas enfermedades (Miklas et al. 2006). Aunque en ocasiones puede resultar más sencillo desarrollar test de resistencia que un análisis de marcadores moleculares, hay casos en que su uso ofrece ventajas como:
- El agrupamiento de genes epistáticos, esto es, que condicionan resistencia a las mismas razas del patógeno (no diferenciables) como el citado ejemplo de los genes Co-2 y Co-9.
- La necesidad de selecciones en fases tempranas, sin que las plantas lleguen a la madurez.
- La selección frente a diferentes patógenos que impida realizar diferentes test de resistencia.
- La selección de genotipos portadores de QTLs cuya identificación exige tediosas evaluaciones.
Por otro lado, el desarrollo y abaratamiento de las técnicas de secuenciación del ADN en los últimos años junto con el desarrollo de herramientas bioinformáticas para analizar toda esta información, ya está permitiendo la secuenciación o resecuenciación del genoma de especies vegetales y cultivares. Así, recientemente ha sido publicada una versión del genoma de judía ensamblado y anotado automáticamente (genotipo G19833) que puede ser consultado en la dirección www.phytozome.net. Esto ya supone un cambio sustancial en el modo de abordar los problemas por los mejoradores vegetales ya que ofrecen nuevas posibilidades como;
- La rápida y precisa identificación de los patógenos mediante la comparación de secuencias (BLAST) de ADN de un aislado y las secuencias disponibles en las bases de datos (GenBank o UNITE database). Obviamente, esta posibilidad depende en gran medida de la calidad de las bases de datos y de la precisa clasificación de los aislados incluidos en ellas. Un ejemplo ha sido la identificación de la especie responsable del oídio y ascochyta en los cultivos de judía del norte de España, Erysiphe polygoni y Phoma exigua var. diversispora, respectivamente.
- La posibilidad de diseñar marcadores moleculares basados en el conocimiento de la secuencia de nucleótidos, marcadores a la carta para cada laboratorio y material. A partir de la secuencia de ADN de un marcador dado se puede, mediante BLAST (Basic Local Alignment Search Tool), identificar la posición genómica y desarrollar otros marcadores en ese entono o bien marcadores moleculares funcionales, es decir marcadores desarrollados a partir de secuencias de exones. De este modo se minimiza el riesgo de recombinación entre el marcador y el carácter de interés. En judía existen publicaciones y bases de datos con marcadores ligados a genes de resistencia (véase por ejemplo lista de SCAR markers en la web de Bean Improvement Cooperative, http://bic.css.msu.edu/).
- Análisis de muchos marcadores a bajo coste y con gran rapidez para la selección genómica. Por ejemplo, mediante la metodología de ‘genotyping by sequencing’ (Elshire et al. 2011) se han obtenido un media de 2500 marcadores SNP (Single Nucleotide Polymorphism) por cromosoma y con una posición física conocida. Este genotipado masivo de una población segregante permite identificar aquellos genotipos portadores de las regiones genómicas de interés y, de este modo, acelerar el proceso de mejora genética. Finalmente, este tipo de análisis ofrece una información valiosa para la diferenciación y protección de las obtenciones vegetales.
Agradecimientos
Los autores agradecen al INIA, Ministerio de Economia y Competitividad, Madrid, la financiación de los trabajos de mejora genética de judía a través de diferentes proyectos de investigación (RTA2009-0093, RTA2011-0076-CO2-01, RTA2012-0056), así como la financiación de un contrato DOC-INIA-CCAA (Ana Campa) y una beca FPI-INIA (Ester Murube).
Bibliografía
- Drijfhout E. 1978. Genetic interaction between Phaseolus vulgaris and bean common mosaic virus with implications for strain identification and breeding for resistance. Agric Res Rep 872, Agric Univ, Wageningen
- Elshire R.J., Glaubitz J.C., Sun Q., Poland J.A., Kawamoto K., Buckler E.S., Mitchell E. 2011. A Robust, Simple Genotyping-by-Sequencing (GBS) Approach for High Diversity. Species. PLoS ONE 6: e19379. doi:10.1371/journal.pone.0019379
- Ferreira J.J., Alvarez E., Fueyo M.A., Roca A., Giraldez R. 2000. Determination of the outcrossing rate of Phaseolus vulgaris L. using seed protein markers. Euphytica 113:259–263
- Ferreira J.J., Campa A., Pérez-Vega E., Rodríguez-Suárez C., Giraldez R. 2012. Introgression and pyramiding into common bean market class fabada of genes conferring resistance to anthracnose and potyvirus. Theor. Appl. Genet. 124:777–788
- Miklas P.N., Kelly J.D., Beebe S.E., Blair M.W. 2006. Common bean breeding for resistance against biotic and abiotic stresses: From classical to MAS breeding. Euphytica 147:105–131
- Moose S.P., Mumm R.H. 2008. Molecular Plant Breeding as the Foundation for 21st Century Crop Improvement. Plant Physiology 147: 969–977
- Petzoldt R, Dickson MH. 1996. Straw test for resistance to white mold in beans. Annu Rep Bean Improv Coop 39:142–143
- Puerta Romero J. 1962. Enfermedades y plagas de la judía. Hojas Divulgadoras N.11. Ministerio de Agricultura, Madrid
- Schwartz H.F., Steadman J.R., Hall R., Forster R.L. 2005. Compendium of bean diseases. H.F The American Phytopathology Society, Minnesota, USA
- Singh S.P., Schwartz H.F. 2010. Breeding common bean for resistance to diseases: a review. Crop Sci 50:2199-2223
- Taylor J.D., Teverson D.M., Davis J.H.C. 1996. Sources of resistance to Pseudomonas syringae pv. phaseolicola races in Phaseolus vulgaris. Plant Pathol. 45:479–485
- Trabanco N., Pérez-Vega E., Campa A., Rubiales D., Ferreira J.J. 2012. Genetic resistance to powdery mildew in common bean. Euphytica 186:875-882
- Van Schoonhoven A, Pastor Corrales MA. 1987. Standard system for the evaluation of bean germplasm. CIAT, Cali Colombia
- Voysest O. 2000. Mejoramiento genético del frijol (Phaseolus vulgaris L.). En: Legado de variedades de América Latina 1930-1999. Centro Internacional de Agricultura Tropical, Cali, Colombia